甲状腺癌(thyroid cancer)是女性第四大常见的恶性肿瘤[1-2],在2017年,全球范围内有56870例新发甲状腺癌病例,占所有新发肿瘤的3.4%[3]。虽然大部分甲状腺癌倾向于生物学及临床的惰性,拥有较好的预后,但仍有少部分甲状腺癌预后差,生存期短以及出现局部及全身转移等,严重威胁人类生命健康[4-6]。由于疾病过程交错复杂,有众多分子参与其中,临床中仍然难以寻找到有效的生物预测因子。侵袭和转移是癌症的一个标志,主要是导致甲状腺癌的复发和预后不良而导致死亡。对于转移及复发性甲状腺癌患者,目前仍缺乏有效治疗手段。因此,深入了解甲状腺癌疾病发生和发展的分子机制,探索新的潜在治疗靶点,将有助于提高患者的生存水平。本研究通过生物信息学方法分析甲状腺癌发生相关差异表达基因,构建其长链非编码RNA(long non-coding RNA,lncRNA)、微小RNA(microRNA,miRNA)及mRNA之间的竞争性内源RNA(competing endogenous RNA,ceRNA)调控网络,并结合TCGA数据库进行预后分析,以期获取影响甲状腺癌预后的关键非编码RNA与mRNA及相关通路。
1 材料与方法
1.1 数据资料收集
在癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库(https://cancergenome.nih.gov)中下载568例甲状腺癌样本并具有完整生存时间的临床数据,使用数据转换工具(GDC Apps提供)下载样本的mRNAseq和miRNAseq的基因表达数据,作为后续核心差异基因的预后分析。
1.2 差异表达基因筛选
应用R程序语言包(http://bioconductor.org/packages/release/bioc/html/edgeR.html)对TCGA数据库下载的mRNAseq和miRNAseq数据信息进行差异表达分析,筛选出甲状腺癌组织和正常组织中差异表达的lncRNA、miRNA及mRNA。差异基因筛选标准:校正后P值<0.01,差异倍数>2.0。
1.3 功能富集及蛋白互作分析
采用Cytoscape v 3.5.1软件对差异表达基因进行GO功能富集分析参与的生物学过程及KEGG通路富集分析。采用Fisher精确检验的方法,以P<0.05及FDR<0.05为显著性基因富集。
1.4 ceRNA调控网络的构建
使用Cytoscape v 3.5.1软件构建差异表达lncRNA、miRNA、mRNA的调控网络。使用miRcode(http://www.mircode.org)预测差异表达lncRNA-miRNA关系对,使用miRTarBase(http://mirtarbase.mbc.nctu.edu.tw),miRDB(http://www.mirdb.org)和TargetScan(http://www.targetscan.org/vert_71)预测差异表达miRNA-mRNA关系对的互作关系。
1.5 差异表达基因的生存分析
将差异表达lncRNA的表达水平进行单因素及多因素Cox回归分析,并将差异表达lncRNA根据中位值分成高水平组和低水平组,应用Kaplan-Meier生存曲线进行生存分析,筛选出与甲状腺癌总生存期显著相关的基因,分析与基因表达与临床参数的关系。使用受试者工作特征曲线(receiver operating characteristic curve,ROC)和ROC曲线面积(AUC),对生存分析预测的敏感性和特异性进行评价。P<0.05表示差异具有统计学意义。
2 结 果
2.1 甲状腺癌的差异表达基因的筛选
在TCGA数据库中下载和提取568例样本的lncRNA数据,数据分为2个队列:510例甲状腺癌样本和58例正常样本。下载和提取573例样本的miRNA数据,数据分为2个队列:514例甲状腺癌样本和59例正常样本。筛选出差异表达lncRNA 497个(上调93个,下调404个),差异表达miRNA 72个(上调5个,下调67个),差异表达mRNA 1097个(上调233个,下调864个)。使用R语言分别制作差异表达的lncRNA、miRNA、mRNA的火山图及热图(图1)。
2.2 差异表达基因的GO富集分析和KEGG通路分析
使用Cytoscape v 3.5.1软件对1097个差异基因进行GO生物过程富集分析和KEGG通路分析。GO生物过程富集分析结果显示差异表达基因主要参与单组织过程、单组织细胞过程、刺激反应等生物学过程,受体结合、分子功能调节、钙离子调节等细胞功能,细胞、细胞膜、细胞外组件等细胞构成过程(图2)。KEGG通路分析结果显示差异表达基因主要参与神经反应受体-配体相互作用、细胞因子-细胞因子受体相互作用、肿瘤转录失调等信号通路的调节(图3)。
2.3 甲状腺癌ceRNA调控网络的构建
使用Cytoscape v 3.5.1软件进行lncRNA-miRNA-mRNA的ceRNA互作网络构建(图4),结果显示有lncRNA-miRNA关系对72对,miRNA-mRNA关系对13对,其中共包括差异表达lncRNA 31个,差异表达miRNA 11个和差异表达mRNA 12个(图4)。
2.4 甲状腺癌的生存分析
在ceRNA互作网络中有2个差异表达lncRNA(MIR181A2HG、OPCML-IT1),1个差异表达miRNA(miRNA-184)和2个差异表达mRNA(E2F1、SALL3)与甲状腺癌的总体生存期明显有关(均P<0.05)。将甲状腺癌样本以中位数值为界值,分成高表达组和低表达组,采用Kaplan-Meier分析两组患者预后相关性,结果表明低表达组具有更长的生存时间(均P<0.05)(图5)。使用ROC曲线和AUC评价5年生存率预测的敏感性和特异性。结果显示,差异表达的lncRNA、miRNA、mRNA的AUC值分别是0.913、0.956、0.955(图6)。
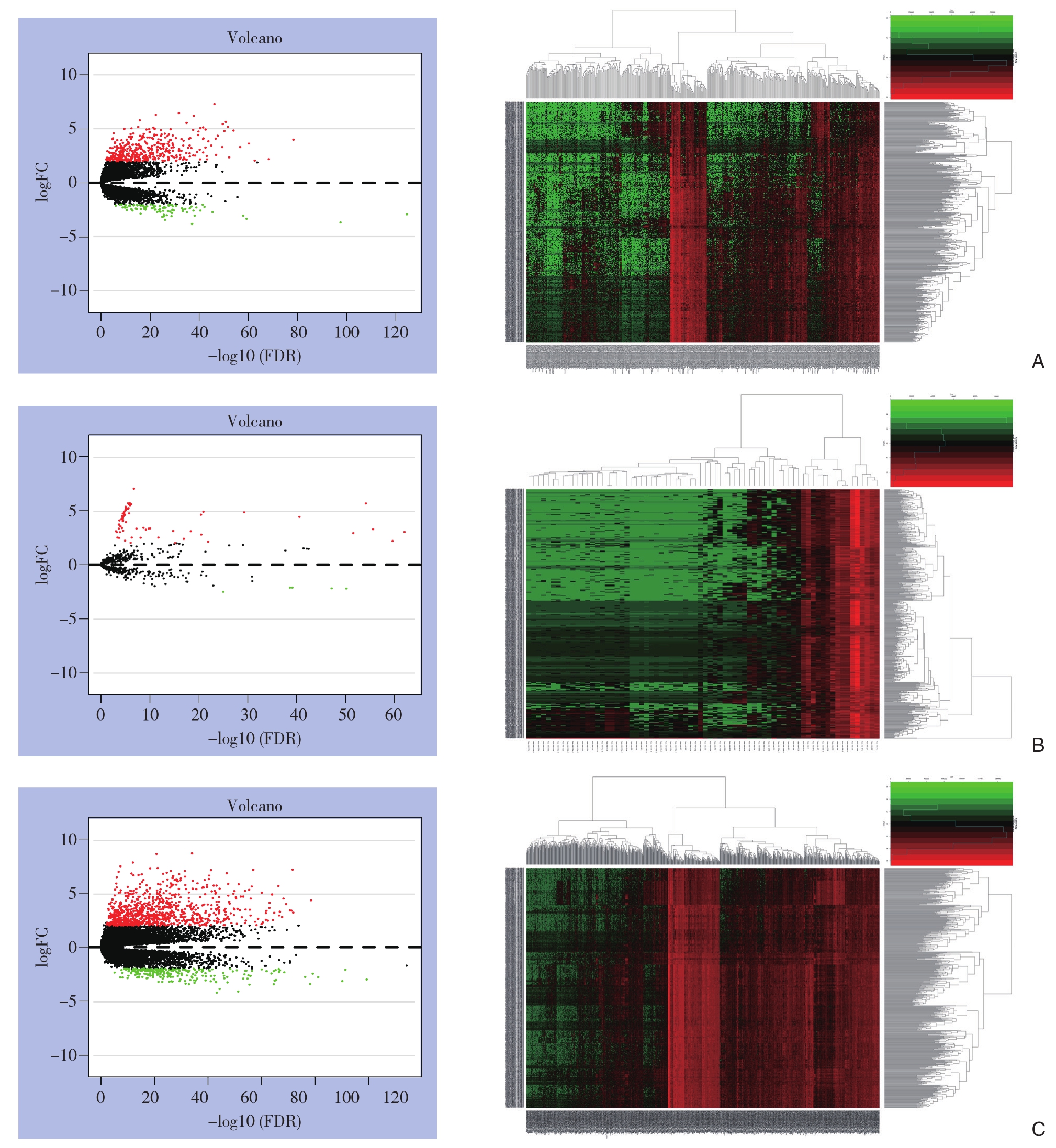
图1 图1 火山图及热图分析 A:差异表达lncRNA;B:差异表达miRNA;C:差异表达mRNA
Figure1 XVolcano plot and heat map analysis A: Differentially expressed lncRNAs; B: Differentially expressed miRNAs; C: Differentially expressed mRNAs
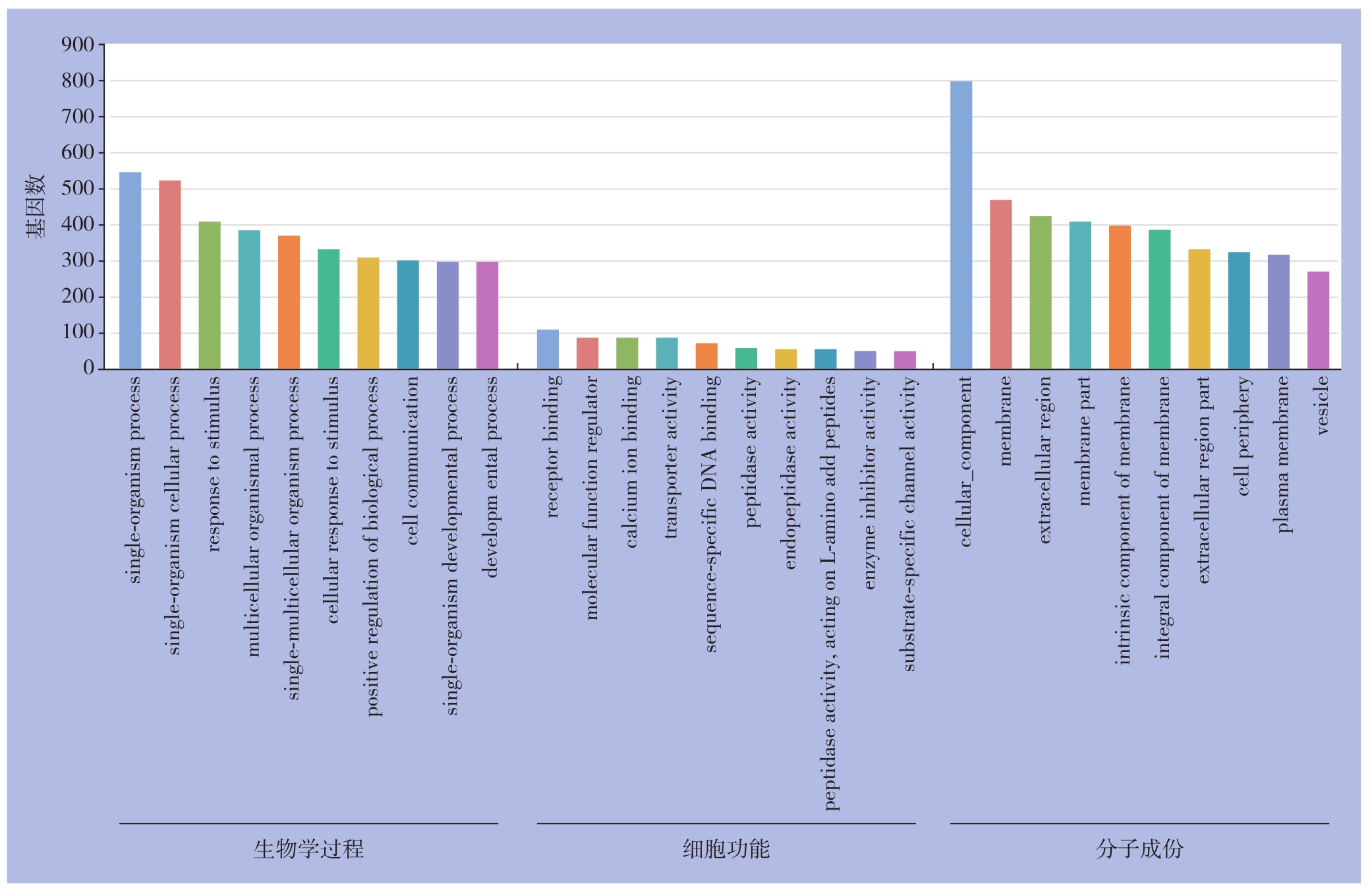
图2 GO功能富集分析
Figure2 GO functional enrichment analysis
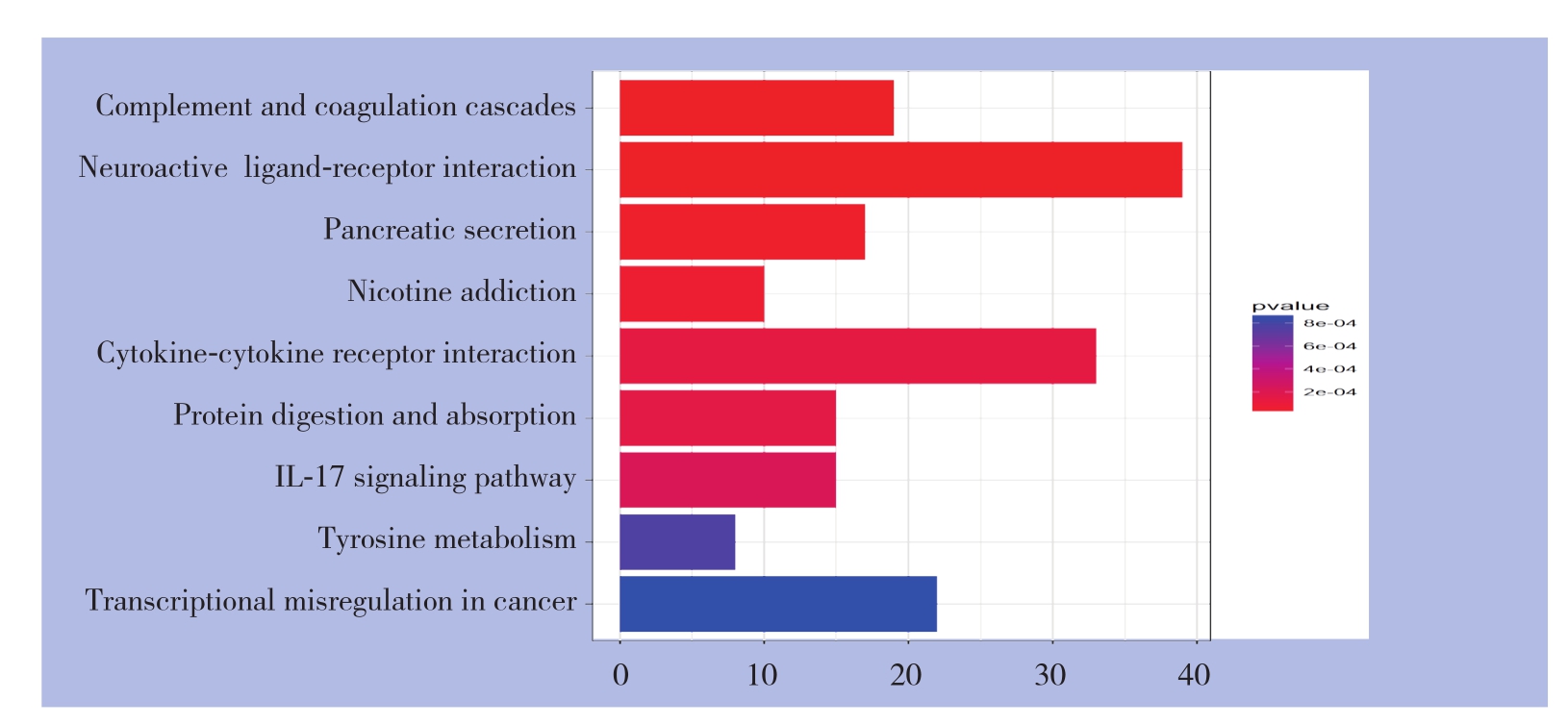
图3 KEGG通路分析
Figure3 KEGG pathway analysis
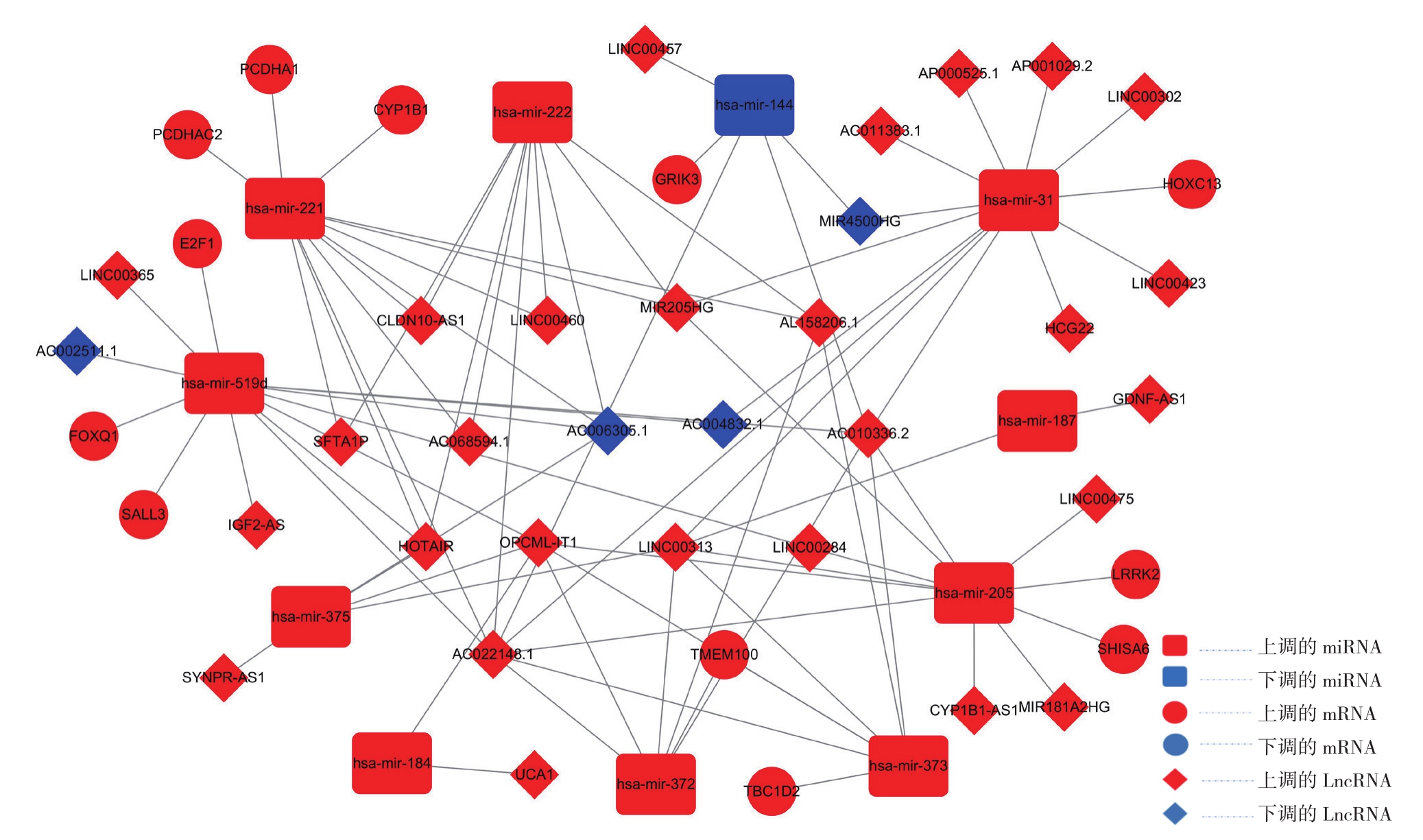
图4 构建LncRNA-miRNA-mRNA(ceRNA)调控网络
Figure4 Construction of regulatory network of lncRNA-miRNA-mRNA (ceRNA)
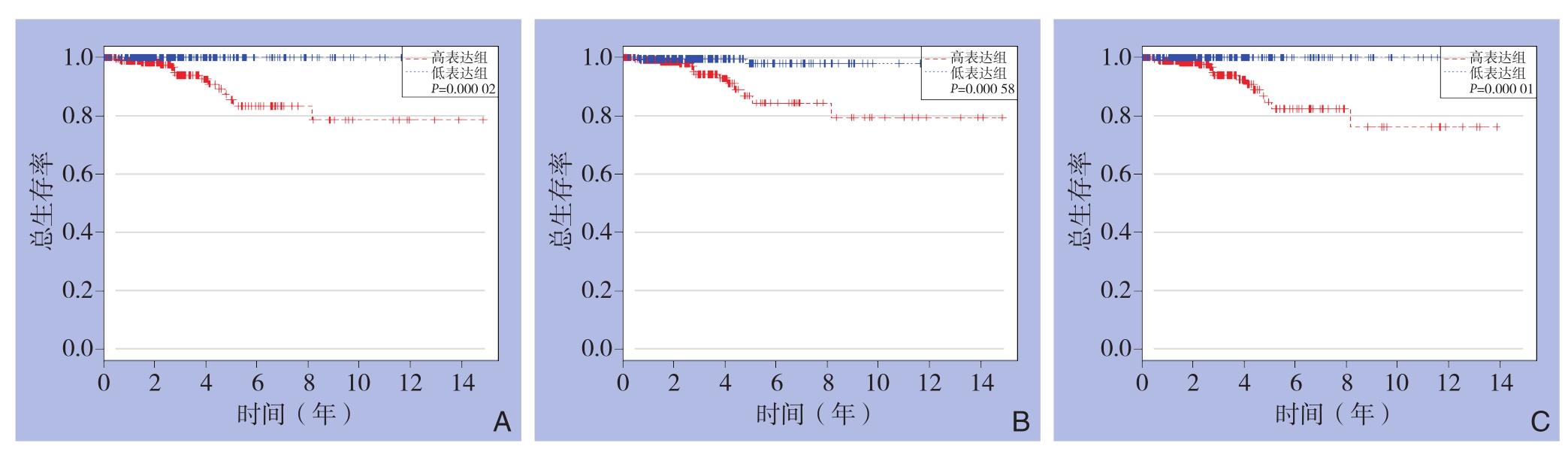
图5 Kaplan-Meier生存曲线 A:差异表达lncRNA;B:差异表达miRNA;C:差异表达mRNA
Figure5 Kaplan-Meier survival curves A: Differentially expressed lncRNAs; B: Differentially expressed miRNAs; C: Differentially expressed mRNAs
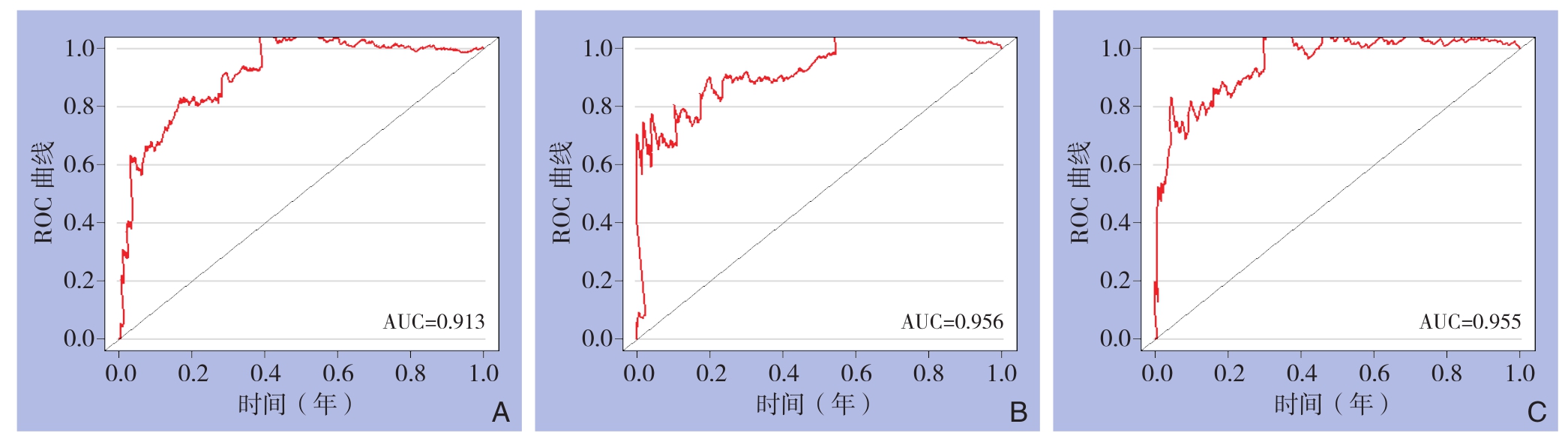
图6 ROC曲线 A:差异表达lncRNA;B:差异表达miRNA;C:差异表达mRNA
Figure6 ROC curves A: Differentially expressed lncRNAs; B: Differentially expressed miRNAs; C: Differentially expressed mRNAs
3 讨 论
lncRNA是一类转录本长度超过200 nt,不编码蛋白的RNA,近年的研究[7-10]表明lncRNA位于细胞核和细胞质中,主要通过表观遗传修饰、转录调控(转录后调控)和翻译水平调控(翻译后调控)发挥作用,也可通过与miRNA或其他细胞因子相互作用,间接影响基因表达。广泛参与包括X染色体沉默、染色质修饰、核内运输等多种重要的调控过程。也有研究[11-12]表明lncRNA的表达变化与功能丧失已经与多种恶性肿瘤在内的人类疾病联系在一起。miRNA是一类转录本长度在20~25 nt的小非编码RNA,miRNA可以抑制mRNA的翻译或引起RNA的转录后翻译的降解[13]。ceRNA假说揭示了一种RNA间相互作用的新机制[14]。lncRNA与miRNA及其下游靶基因之间的相互调控模式与肿瘤的发生发展密切相关,成为肿瘤研究领域的热点。miRNA作为一个转录后调控的重要因子,其活性可被lncRNA通过“海绵”吸附的方式调控ceRNA[15]。lncRNA作为ceRNA竞争性地与miRNA结合从而影响miRNA导致的基因沉默,从而调节编码基因的蛋白质水平,参与靶基因的表达调控,在肿瘤的发生发展中发挥重要的作用。ceRNA构建了一个复杂的作用网络,正常生理状态下ceRNA网络中的各种分子之间处于一定的平衡状态,一旦平衡被打破就会导致疾病的发生。目前有研究揭示了ceRNA调控网络在部分肿瘤中的主要作用[16-18]。目前仍缺乏在全基因组范围内,特别是基于高通量检测与大规模样本量的甲状腺癌相关的lncRNA和miRNA及的ceRNA的综合分析。
MIR181A2HG(MIR181A2 host gene,MIR181A2宿主基因)是位于染色体9q33.3的lncRNA,miR-181a 从两条人类染色体的lncRNA转录而来,分别是1号染色体的MIR181A1HG和9号染色体的MIR181A2HG。有报道MIR181A2HG可以促进子宫相关炎症通路的发生[19]。OPCMLIT1(OPCML intronic transcript 1,OPCML内转录1)是位于11号染色体的lncRNA。目前尚无MIR181A2HG和OPCML-IT1在甲状腺癌中的表达及作用的相关报道,有待于我们进一步进行基础实验和临床试验。miR-184近年来作为一个新的miRNA在许多肿瘤中表现出异常的表达,包括肝细胞癌(作为肝细胞癌的致癌调节剂)和肾细胞癌(作为肿瘤抑制因子)等[20-21]。E2F1是炎症控制的关键参与者,它与成视网膜细胞瘤蛋白(pRb)息息相关。pRb通过对E2F家族转录因子的诱导来控制细胞周期。有研究[22]表明变异的pRb通过激活E2F1来抑制甲状腺肿瘤。Sall3(spalt like transcription factor 3)编码C2H2锌指蛋白,这种蛋白质存在于进化保守的基因家族中,包括果蝇、新杆状线虫和脊椎动物[23]。近期有研究[24]表明SALL3的表达与致癌作用之间有关系。研究表明在肝细胞癌中SALL3被DNA甲基化所抑制,其蛋白质与DNA甲基转移酶3α(DNMT3A)相互作用。另一个研究[25]则指出SALL3和HPV感染的异常超甲基化对宫颈癌的致癌作用有促进作用。SALL3在甲状腺癌中是否有促癌作用尚未见报道。
通过对lncRNA和miRNA的靶基因预测和功能分析发现,GO生物过程富集分析结果显示差异表达基因主要参与单组织过程、单组织细胞过程、刺激反应等生物学过程,受体结合、分子功能调节、钙离子调节等细胞功能,细胞、细胞膜、细胞外组件等细胞构成过程。KEGG通路分析结果显示差异表达基因主要参与神经反应受体-配体相互作用、细胞因子-细胞因子受体相互作用、肿瘤转录失调等信号通路的调节。因此,初步探讨与预后相关的lncRNA和miRNA的靶基因生物过程富集和信号通路分析,对于揭示甲状腺癌发生和发展的分子机制以及肿瘤的诊断治疗具有一定的临床意义。
本研究对T C G A数据库中甲状腺癌的lncRNA,miRNA和mRNA测序数据与甲状腺癌的临床信息进行了相关的统计学分析。其优势在于研究样本量大,测试平台统一,结果全面可靠。但其结果具有一定局限性,需要在基础实验和临床试验中进一步验证。
综上所述,通过对甲状腺癌患者的lncRNA,miRNA和mRNA数据进行分析发现,差异表达lncRNA(MIR181A2HG、OPCML-IT1),差异表达miRNA(miRNA-184)和差异表达mRNA(E2F1、SALL3)可能在甲状腺癌的发生发展中发挥作用,具有成为新的预测甲状腺癌预后分子标志物的潜能。对于揭示甲状腺癌发生、发展的分子机制及肿瘤的诊断和分子靶向治疗具有一定的临床意义。
[1] National Cancer Institute. SEER Cancer Statistics Review 1975–2009 (Vintage 2009 Populations)[DB]. http://seer.cancer.gov/csr/1975_2009_pops09/browse_csr.php?section=28&page=sect_28_table.01.html.
[2] Udelsman R, Zhang Y. The epidemic of thyroid cancer in the United States: The role of endocrinologists and ultrasounds[J]. Thyroid,2014, 24(3):472–479. doi: 10.1089/thy.2013.0257.
[3] Yapa S, Mulla O, Green V, et al. The Role of Chemokines in Thyroid Carcinoma[J]. Thyroid, 2017, 27(11):1347–1359. doi:10.1089/thy.2016.0660.
[4] Nikiforov YE, Nikiforova MN. Molecular genetics and diagnosis of thyroid cancer[J]. Nat Rev Endocrinol, 2011, 7(10):569–580. doi:10.1038/nrendo.2011.142.
[5] Livolsi VA. Papillary thyroid carcinoma: an update[J]. Mod Pathol,2011, 24(Suppl 2):S1–9. doi: 10.1038/modpathol.2010.129.
[6] 王俊男, 闫枫尚, 徐拯, 等. 基于SEER数据库的甲状腺髓样癌预后因素分析[J]. 中国普通外科杂志, 2018, 27(5):547–552.doi:10.3978/j.issn.1005–6947.2018.05.004.Wang JN, Yan FS, Xu Z, et al. Analysis of prognostic factors for medullary thyroid carcinoma based on SEER database[J]. Chinese Journal of General Surgery, 2018, 27(5):547–552. doi:10.3978/j.issn.1005–6947.2018.05.004.
[7] Jalali S, Bhartiya D, Lalwani MK, et al. Systematic transcriptome wide analysis of lncRNA-miRNA interactions[J]. PLoS One, 2013,8(2):e53823. doi: 10.1371/journal.pone.0053823.
[8] Ye S, Yang L, Zhao X, et al. Bioinformatics method to predict two regulation mechanism: TF-miRNA-mRNA and lncRNA-miRNA-mRNA in pancreatic cancer[J]. Cell Biochem Biophys, 2014,70(3):1849–1858. doi: 10.1007/s12013–014–0142-y.
[9] Lopes AM, Arnold-Croop SE, Amorim A, et al. Clustered transcripts that escape X inactivation at mouse XqD[J]. Mamm Genome, 2011,22(9/10):572–582. doi: 10.1007/s00335–011–9350–6.
[10] Kallen AN, Zhou XB, Xu J, et al. The imprinted H19 lncRNA antagonizes let-7 microRNAs[J]. Mol Cell, 2013, 52(1):101–112.doi: 10.1016/j.molcel.2013.08.027.
[11] Huarte M, Guttman M, Feldser D, et al. A large intergenic noncoding RNA induced by p53 mediates global gene repression in the p53 response[J]. Cell, 2010, 142(3):409–419. doi: 10.1016/j.cell.2010.06.040.
[12] Malik R, Patel L, Prensner JR, et al. The lncRNA PCAT29 inhibits oncogenic phenotypes in prostate cancer[J]. Mol Cancer Res, 2014,12(8):1081–1087. doi: 10.1158/1541–7786.MCR-14–0257.
[13] Macfarlane LA, Murphy PR. MicroRNA: Biogenesis, Function and Role in Cancer[J]. Curr Genom, 2010, 11(7):537–561. doi:10.2174/138920210793175895.
[14] Salmena L, Poliseno L, Tay Y, et al. A ceRNA hypothesis:the Rosetta Stone of a hidden RNA language?[J]. Cell, 2011,146(3):353–358. doi: 10.1016/j.cell.2011.07.014.
[15] Ballantyne MD, McDonald RA, Baker AH. LncRNA/MicroRNA interactions in the vasculature[J]. Clin Pharmacol Ther, 2016,99(5):494–501. doi: 10.1002/cpt.355.
[16] Cheng DL, Xiang YY, Ji LJ, et al. Competing endogenous RNA interplay in cancer: mechanism, methodology, and perspectives[J].Tumour Biol, 2015, 36(2):479–488. doi: 10.1007/s13277–015–3093-z.
[17] Sen R, Ghosal S, Das S, et al. Competing endogenous RNA: the key to posttranscriptional regulation[J]. Scient World J, 2014,2014:896206. doi: 10.1155/2014/896206.
[18] Qi X, Zhang DH, Wu N, et al. ceRNA in cancer: possible functions and clinical implications[J]. J Med Genet, 2015, 52(10):710–718.doi: 10.1136/jmedgenet-2015–103334.
[19] Gao L, Wang G, Liu WN, et al. Reciprocal Feedback Between miR-181a and E2/ERα in Myometrium Enhances Inflammation Leading to Labor[J]. J Clin Endocrinol Metab, 2016,101(10): 3646–3656.doi: 10.1210/jc.2016–2078.
[20] Su Z, Chen D, Li Y, et al. microRNA-184 functions as tumor suppressor in renal cell carcinoma[J]. Exp Ther Med, 2015,9(3):961–966. doi: 10.3892/etm.2015.2199.
[21] Gao B, Gao K, Li L, et al. miR-184 functions as an oncogenic regulator in hepatocellular carcinoma (HCC) [J].Biomed Pharmacother, 2014, 68(2):143–148. doi: 10.1016/j.biopha.2013.09.005.
[22] Toki H, Inoue M, Minowa O, et al. Novel retinoblastoma mutation abrogating the interaction to E2F2/3, but not E2F1, led to selective suppression of thyroid tumors[J]. Cancer Sci, 2014, 105(10):1360–1368. doi: 10.1111/cas.12495.
[23] Sweetman D, Munsterberg A. The vertebrate spalt genes in development and disease[J]. Dev Biol. 2006, 293(2):285–293. doi:10.1016/j.ydbio.2006.02.009.
[24] Shikauchi Y, Saiura A, Kubo T, et al. SALL3 interacts with DNMT3A and shows the ability to inhibit CpG island methylation in hepatocellular carcinoma[J]. Mol Cell Biol, 2009, 29(7):1944–1958. doi: 10.1128/MCB.00840–08.
[25] Wei X, Zhang S, Cao D, et al. Aberrant Hypermethylation of SALL3 with HPV Involvement Contributes to the Carcinogenesis of Cervical Cancer[J]. PLoS One, 2015, 10(12):e0145700. doi:10.1371/journal.pone.0145700.
