原发性肝细胞癌(hepatocellular carcinoma,HCC)是全球最常见的恶性肿瘤之一,其发病率居世界第6位,致死率居世界第2位[1]。现阶段,尽管HCC的诊疗技术较前已有一定的提高,但HCC早期诊断困难、易复发、易转移等特点仍使得HCC患者的整体预后欠佳[2]。因此,深入研究HCC潜在的发生、发展机制,对于提升HCC的诊治水平以及改善患者的预后有着重要意义。
微小RNA(microRNA,miRNA)是一类长度约为22个核苷酸的内源性非编码RNA分子,其可在转录后水平与mRNA特异性结合下调靶基因的表达。诸多研究[3-4]表明,miRNA的异常表达与肿瘤的发生、发展及预后密切相关,具有癌基因或抑癌基因的功能。已有研究发现,miR-1180在多种实体肿瘤中存在异常表达,且呈现出一定的组织或肿瘤特异性。例如,miR-1180在肺癌和胰腺癌中表达上调,能促进癌细胞的增殖、迁移和侵袭,并与肺癌患者TNM分期和不良预后密切相关;但miR-1180在膀胱癌中却呈现低水平表达,发挥着抑癌基因的作用[5-7]。目前,miR-1180与HCC的相关性研究鲜有报道,故本研究基于GEO(Gene Expression Omnibus)和TCGA(The Cancer Genome Atlas)数据库来探讨miR-1180在HCC组织中的表达情况及其临床意义,并对miR-621靶基因进行系统的生物信息学分析,以期为寻找新型HCC诊断标志物或治疗靶点提供依据。
1 资料与方法
1.1 数据来源
本研究自GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)下载了数据集GSE36915 miRNA表达数据,包含68例HCC组织,21例癌旁组织。从TCGA数据库(https://cancergenome.nih.gov/)中下载了377例HCC组织样本和50例癌旁组织样本的miRNAseq、mRNAseq表达谱和临床数据(3级数据),其中371例患者具有匹配的miRNA表达谱和临床数据,365例患者具有匹配的miRNA表达谱、mRNA表达谱和临床数据。
1.2 方法
1.2.1 差异表达分析 利用R(The Project for Statistical Computing)edgeR 包 [8](http://www.bioconductor.org/packages/release/bioc/html/edgeR.html)分别对miRNA和mRNA表达谱进行过滤、归一化处理,筛选HCC组织与癌旁组织之间差异表达mRNA和miRNA(筛选标准:|log2差异倍数 |>1,P<0.05)。标准化后的 miRNA 和mRNA表达值以每100万标记读本中外显子的读本数(counts of exon model per million mapped reads,CPM)表示,并经log2转化后用于后续分析。
1.2.2 患者分组 提取TCGA数据库371例HCC患者匹配的miR-1180表达谱和预后数据,使用X-tile3.6.1(Yale University School of Medicine,New Haven,CT,USA)软件 [9]基于Kaplan-Meier法及Log-rank检验确立最佳截断值7.05,将患者分为miR-1180低表达组(n=250)和miR-1180高表达组(n=121)。
1.2.3 miR-1180靶基因预测及功能分析 使用 DIANA-microT(http:// diana.imis.athenainnovation.gr)、TargetScan v7.1(http://www.targetscan.org)、MiDRB(http://www.mirdb.org/)3个数据库联合(取交集)预测miR-1180靶基因;利用miRwalk(http://129.206.7.150/)、Tarbase(http://carolina.imis.athena-innovation.gr)、miRTraBase(http://mirtarbase.mbc.nctu.edu.tw)3个数据库检索(取并集)已被实验所证实的miR-1180靶基因。两者的合集与HCC组织中差异低表达基因集合的交集即为本研究所确立的miR-1180靶基因集合。进而,利用在线 工 具 DAVID(https://david.ncifcrf.gov/tools.jsp)对miR-1180靶基因进行基因本体论(Gene ontology,GO) 及 KEGG(Kyoto encyclopedia of genes and genomes)通路富集分析。GO富集分析包括生物过程(biological process,BP)、细胞成分(cellular component,CC)和分子功能(molecular function,MF)3个部分。
1.2.4 关键靶基因筛选 利用在线工具STRING[10](https://string-db.org)联合Cytoscape软件[11]构建miR-1180靶基因蛋白互作网络,并运用CytoHubba插件依据连接度(degree)筛选出前30位关键基因(hub gene)。提取365例HCC患者关键基因的表达谱及预后数据,采用X-tile 3.6.1软件筛选预后风险基因,确立最佳截断值并绘制Kaplan-Meier曲线。
1.3 统计学处理
采用SPSS 22.0软件进行统计分析。计量资料采用独立样本t检验。受试者工作特征曲线(ROC)用于评价指标诊断效能,计算曲线下面积(area under the curve,AUC)。生存分析采用Kaplan-Meier法及Log-rank检验,并运用Cox比例风险回归模型分析影响患者预后的危险因素,计算风险比(hazard ratio,HR)及其95%可信区间(confidence interval,CI)。结局指标采用患者总生存期(overall Survival,OS),即术后病理确诊之日起至患者因任何原因死亡的间隔时间。以P<0.05为差异具有统计学意义。
2 结 果
2.1 HCC组织与癌旁组织miR-1180差异表达分析
GSE36915数据集与TCGA数据集的分析结果一致显示miR-1180在HCC组织中较癌旁组织差异高表达(均P<0.0001),且对于HCC具有良好的诊断效能(GSE36915:AUC=0.8 704,P<0.0 001;TCGA:AUC=0.8 450,P<0.0 001)(图1)。

图1 miR-1180在HCC组织及癌旁组织中表达量及其诊断HCC的ROC曲线
Figure 1 Expression levels of miR-1180 in HCC tissues and adjacent liver tissues, and ROC curves of miR-1180 for diagnosis of HCC
2.2 miR-1180表达与HCC患者临床特征的关系
回顾性分析371例HCC患者miR-1180表达量与临床特征关系,结果显示miR-1180的表达水平与患者年龄、肿瘤家族史、肿瘤分化程度、AFP等密切相关(均P<0.05),而与性别、TNM分期、纤维化评分、Child-Pugh分级、血管侵犯等指标无明显关系(均P>0.05)(表1)。
表1 HCC患者miR-1180表达量与临床病理参数之间的关系
Table 1 Relations of miR-1180 expression with clinicopathologic factors of the HCC patients
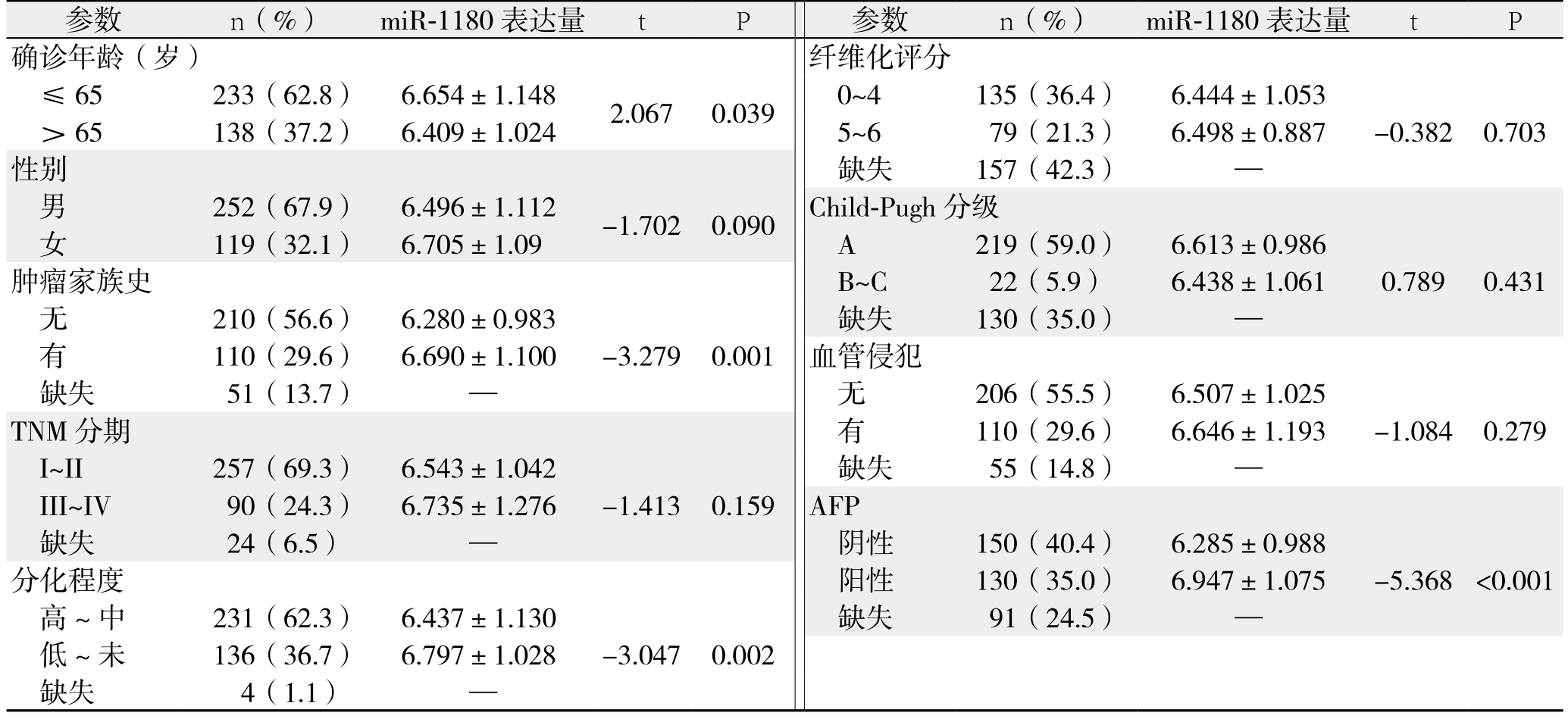
参数 n(%) miR-1180表达量 t P 参数 n(%) miR-1180表达量 t P确诊年龄(岁) 纤维化评分≤65 233(62.8) 6.654±1.148 2.067 0.039 0~4 135(36.4) 6.444±1.053>65 138(37.2) 6.409±1.024 5~6 79(21.3) 6.498±0.887 -0.382 0.703性别 缺失 157(42.3) —男252(67.9) 6.496±1.112 -1.702 0.090 Child-Pugh 分级女119(32.1) 6.705±1.09 A 219(59.0) 6.613±0.986肿瘤家族史 B~C 22(5.9) 6.438±1.061 0.789 0.431无210(56.6) 6.280±0.983-3.279 0.001缺失 130(35.0) —有110(29.6) 6.690±1.100 血管侵犯缺失 51(13.7) — 无 206(55.5) 6.507±1.025 TNM分期 有 110(29.6) 6.646±1.193 -1.084 0.279 I~II 257(69.3) 6.543±1.042 缺失 55(14.8) —III~IV 90(24.3) 6.735±1.276 -1.413 0.159 AFP缺失 24(6.5) — 阴性 150(40.4) 6.285±0.988分化程度 阳性 130(35.0) 6.947±1.075 -5.368 <0.001高~中 231(62.3) 6.437±1.130 缺失 91(24.5) —低~未 136(36.7) 6.797±1.028 -3.047 0.002缺失 4(1.1) —
2.3 HCC患者预后影响因素分析
Kaplan-Meier分析提示HCC组织中miR-1180高表达涉及HCC患者不良预后(P=0.0 012)(图2)。此外,单因素Cox回归分析表明miR-1180表达量、TNM分期、血清甲胎蛋白与HCC患者OS密切有关(均P<0.05);多因素Cox回归分析则进一步提示TNM分期(HR=1.734,95% CI=1.127~2.668,P=0.012)、miR-1180表达量(HR=1.692,95% CI=1.152~2.485,P=0.007)是影响HCC患者OS的独立危险因素(表2)。
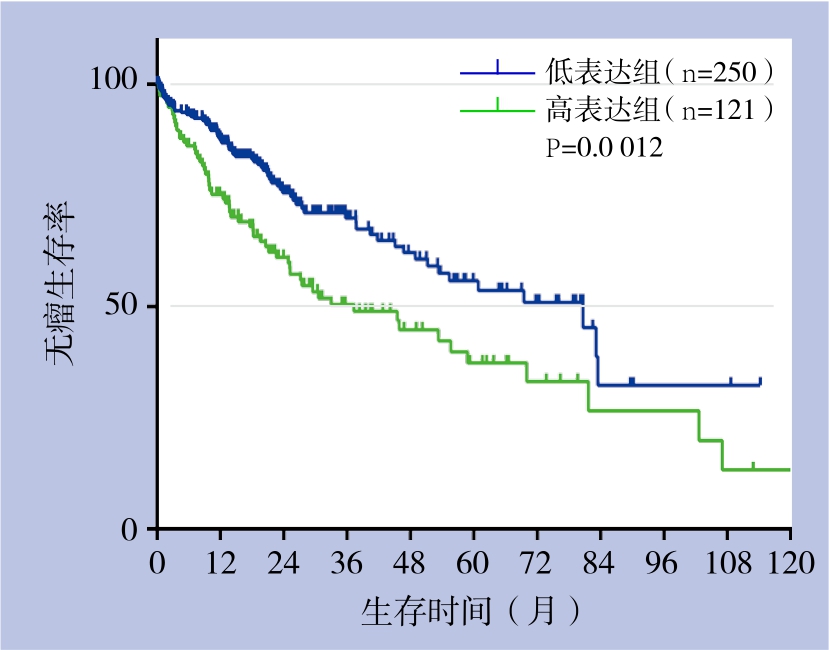
图2 不同miR-1180表达量HCC患者的生存曲线
Figure 2 Survival curves of HCC patients with different miR-1180 expression levels
表2 HCC患者总体生存率影响因素的Cox回归分析
Table 2 Cox regression analysis of factors affecting overall survival in HCC patients
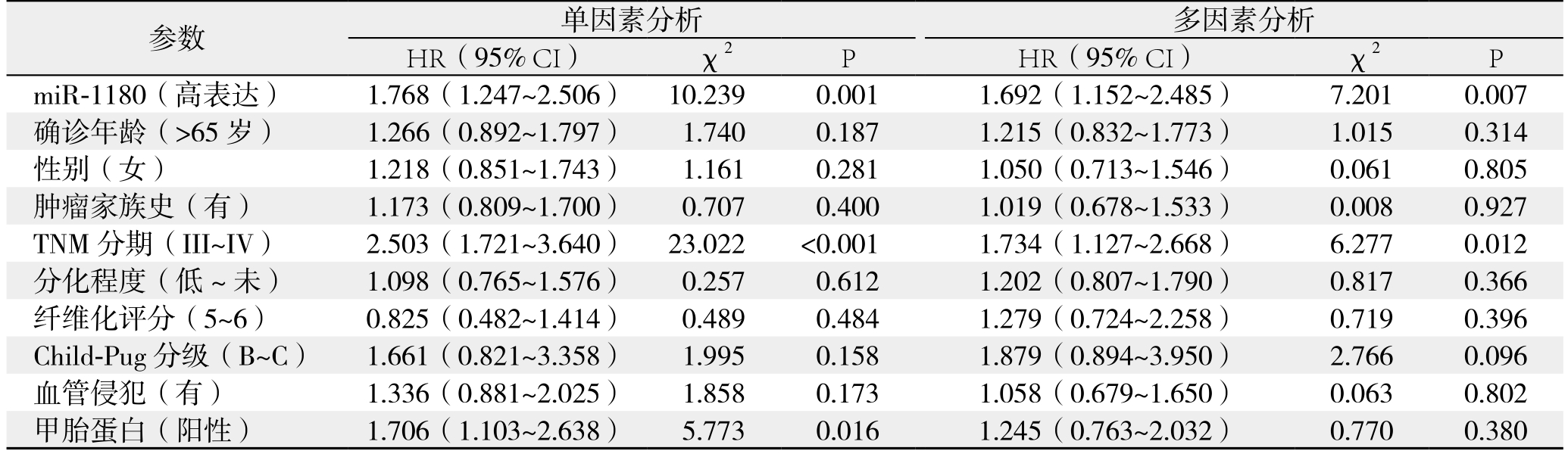
参数 单因素分析 多因素分析HR(95% CI) χ2 P HR(95% CI) χ2 P miR-1180(高表达) 1.768(1.247~2.506) 10.239 0.001 1.692(1.152~2.485) 7.201 0.007确诊年龄(>65岁) 1.266(0.892~1.797) 1.740 0.187 1.215(0.832~1.773) 1.015 0.314性别(女) 1.218(0.851~1.743) 1.161 0.281 1.050(0.713~1.546) 0.061 0.805肿瘤家族史(有) 1.173(0.809~1.700) 0.707 0.400 1.019(0.678~1.533) 0.008 0.927 TNM分期(III~IV) 2.503(1.721~3.640) 23.022 <0.001 1.734(1.127~2.668) 6.277 0.012分化程度(低~未) 1.098(0.765~1.576) 0.257 0.612 1.202(0.807~1.790) 0.817 0.366纤维化评分(5~6) 0.825(0.482~1.414) 0.489 0.484 1.279(0.724~2.258) 0.719 0.396 Child-Pug分级(B~C) 1.661(0.821~3.358) 1.995 0.158 1.879(0.894~3.950) 2.766 0.096血管侵犯(有) 1.336(0.881~2.025) 1.858 0.173 1.058(0.679~1.650) 0.063 0.802甲胎蛋白(阳性) 1.706(1.103~2.638) 5.773 0.016 1.245(0.763~2.032) 0.770 0.380
2.4 miR-1180靶基因预测及功能分析
miR-1180有两种成熟体形式:miR-1180-3p和miR-1180-5p,分别来源于miR-1180前体的3'端和5'端。本研究共筛选获得miR-1180靶基因169个,其中miR-1180-3p靶基因39个,miR-1180-5p靶基因137个。富集分析提示miR-1180-3p的靶基因主要富集于脂质运输、上皮细胞迁移调控等生物过程(BP),低密度脂蛋白颗粒结合等分子功能(MF),以及乙醛酸和二羧酸代谢通路(KEGG)。miR-1180-5p主要富集于细胞雌二醇刺激应答、细胞迁移调控等生物过程(BP),肝素结合、钙离子结合等分子功能(MF),以及缬氨酸、亮氨酸和异亮氨酸降解、补体级联、脂肪酸降解等通路(KEGG)(图3)。
2.5 miR-1180关键靶基因筛选
结合蛋白互作网络和预后分析结果,本研究共筛选出6个miR-1180关键靶基因:过氧化物酶增殖激活受体γ辅激活蛋白1α(peroxisome proliferator activated receptor γ coactivator 1 α,PPARGC1A)、乙醛脱氢酶2(aldehyde dehydrogenase 2 family,ALDH2)、肌氨酸脱氢酶(sarcosine dehydrogenase,SARDH)、3-羟基-3-甲基戊二酰辅酶A合成酶2(3-hydroxy-3-methylglutaryl-CoA synthase 2,HMGCS2)、雌激素受体1(estrogen receptor 1,ESR1)、E26转录因子2(ETS proto-oncogene 1 transcription factor,ETS2);其中,ETS2为miR-1180-3p的靶基因;PPARGC1A、ALDH2、SARDH、HMGCS2、ESR1为miR-1180-5p的靶基因。上述6个基因在HCC组织中均呈现表达下调,且相对低表达者与不良预后密切相关(均P<0.05);比对miR-1180-3p/5p及上述靶基因序列,证实了miR-1180-3p/5p可与相应靶基因3'端非编码区(3'-UTR)结合(图4-5)。
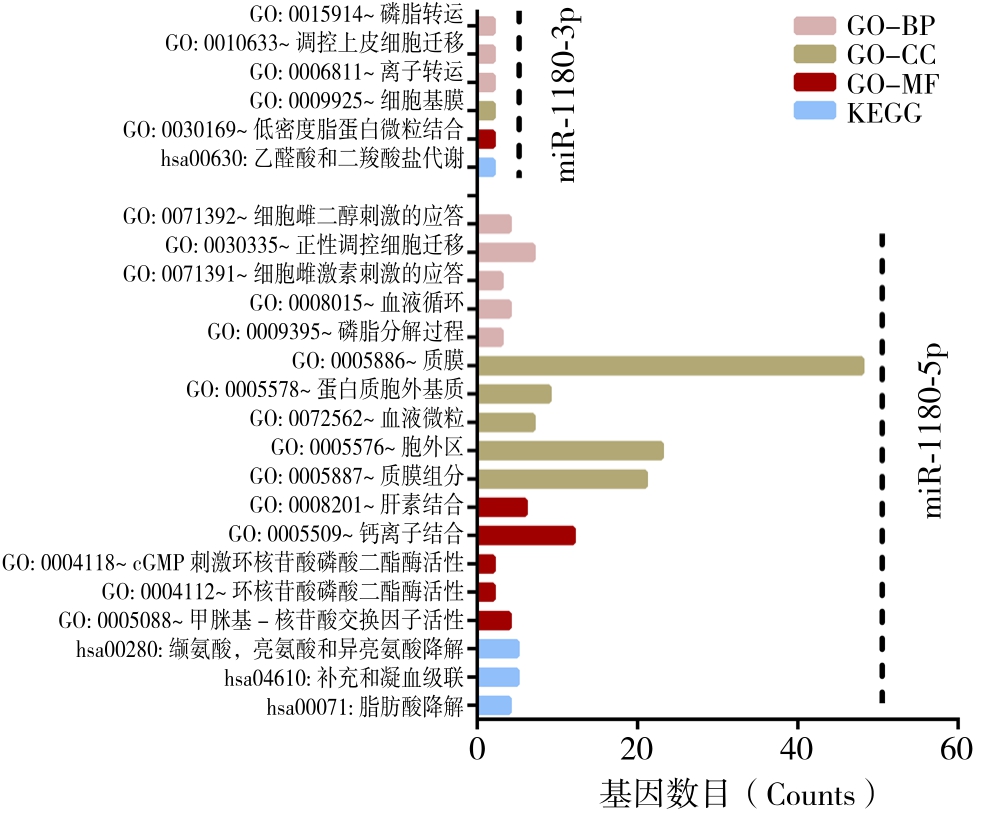
图3 miR-1180靶基因的功能及通路富集分析
Figure 3 Functional and pathway enrichment analyses of the target genes of miR-1180
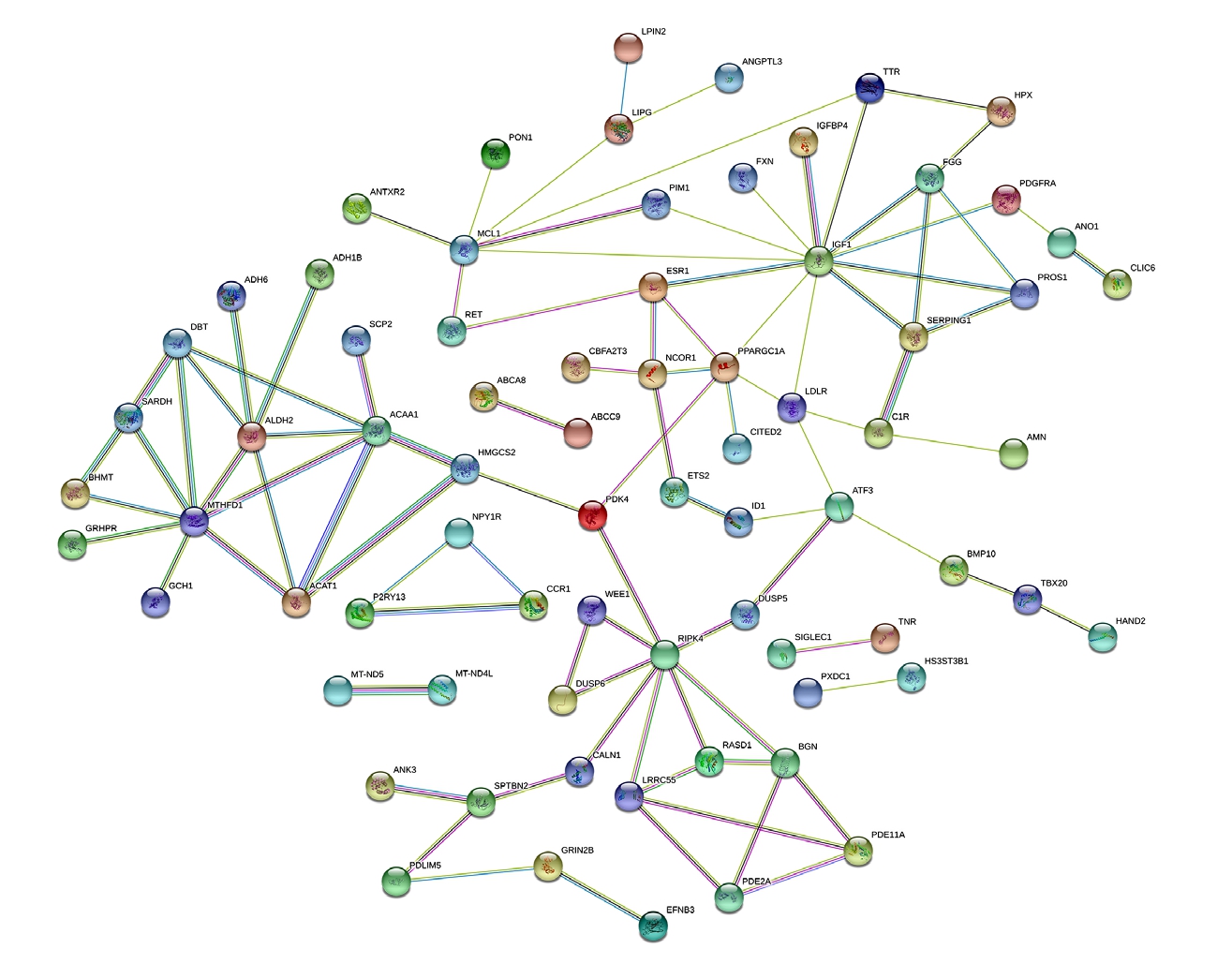
图4 miR-1180靶基因的蛋白互作网络图
Figure 4 The protein-protein interaction networks of the target genes of miR-1180
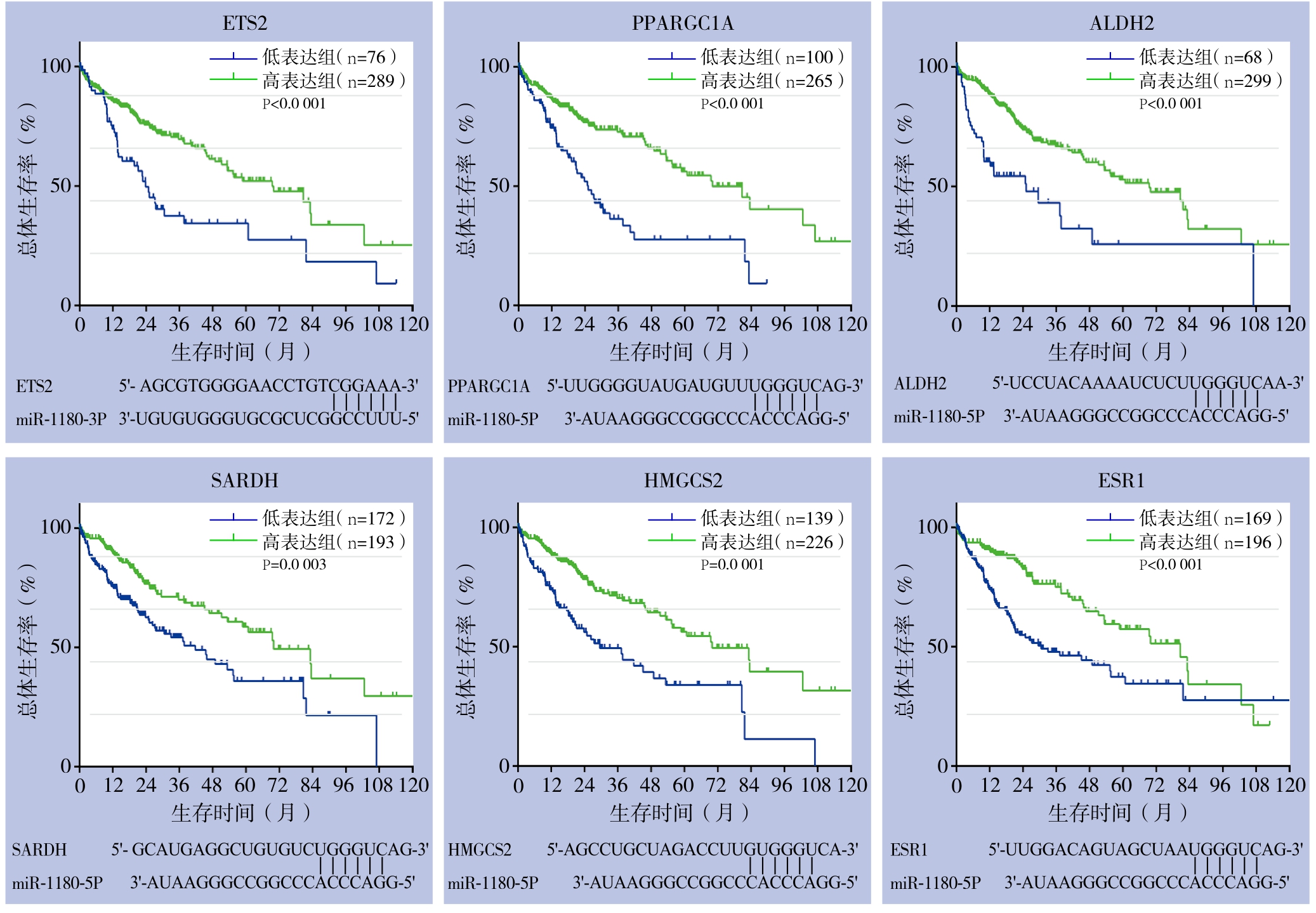
图5 miR-1180关键靶基因相关的生存曲线及miR-1180与其靶基因在3'-UTR的结合位点
Figure 5 Survival curves associated with the key target genes of miR-1180 and the binding sites of miR-1180 in the 3’-UTR of the target genes
3 讨 论
miRNA表达异常在肿瘤的发生、发展中发挥着重要作用,深入研究miRNA与肿瘤的相关性将有望为肿瘤的诊断及治疗提供新思路和新策略。已有研究发现,miR-1180在多种肿瘤组织或肿瘤细胞株中存在异常表达,且呈现出一定的组织或肿瘤特异性,发挥着癌基因(如肺癌、胰腺癌等)或抑癌基因(如膀胱癌等)的作用。例如,肺癌组织中高水平表达miR-1180与患者TNM分期及预后显著相关[5];胰腺癌细胞中过表达miR-1180能下调靶基因TNIP2的表达,激活NF-κB信号通路,促进癌细胞的增殖、迁移和侵袭能力[6];而膀胱癌细胞中miR-1180与抑癌基因P21均呈现低表达,若过表达miR-1180则能显著上调P21表达水平,抑制膀胱癌细胞的增殖[7]。因此,深入探讨miR-1180在不同肿瘤中的作用及其机制有着非常重要的意义。
目前,对于miR-1180在HCC组织中的表达水平及其作用尚不十分明确。Zhou 等[12-13]研究发现HCC组织(8例)及HepG2等HCC细胞株中miR-1180的表达水平较其癌旁组织及LO2正常肝细胞显著上调,且过表达miR-1180可显著下调其靶基因TNIP2、OTUD7B的表达,激活NF-κB信号通路,显著增强癌细胞增殖、迁移和侵袭能力。考虑到上述研究存在样本量较少等不足,本研究首先基于GEO及TCGA数据库的大样本优势分析了miR-1180在HCC组织中的表达情况,证实了miR-1180在HCC组织中的表达水平较癌旁组织显著上调;另外,ROC曲线结果显示miR-1180表达量能较好地区分HCC组织和癌旁组织,提示了miR-1180具有作为HCC病理诊断标志物的潜在价值。
此外,本研究进一步探讨了HCC组织中miR-1180表达量与HCC患者临床病理指标及预后间的相关性。本研究发现miR-1180在低或未分化HCC组织中表达相对较高,且miR-1180表达量与肿瘤家族史相关,这提示了miR-1180密切涉及HCC的发生与发展。值得注意的是,本研究还发现HCC组织中miR-1180表达量与HCC患者血清甲胎蛋白表达呈正相关。已有研究证实,多种miRNA分子在肿瘤患者癌组织及血清中可同时存在表达异常,且具备一定的稳定性和组织特异性,例如miR-215[14]、miR-329[15]等。目前,笔者暂未发现miR-1180在HCC患者血清中表达量的相关研究,对于miR-1180能否成为一种新型HCC血清分子诊断标志物,以及能否联合AFP用于HCC患者的早期诊断有待进一步的研究阐明。此外,Kaplan-Meier分析提示HCC组织中高表达miR-1180与HCC患者不良预后有关,且多因素Cox回归分析证实miR-1180表达量是影响HCC患者预后的独立危险因素。因此,结合miR-1180的表达水平及其与临床病理指标和预后的相关性,推测miR-1180在HCC的发生、发展过程中主要发挥癌基因的作用。
基于miRNA-mRNA互作原理以及系统的生物信息学方法,我们还首次对miR-1180的靶基因进行了较为全面的预测并功能分析。富集分析提示miR-1180的靶基因主要富集于脂质代谢、细胞迁移、转录调控、细胞信号转导等功能,以及脂肪酸降解及缬氨酸、亮氨酸和异亮氨酸降解等通路。诸多研究表明,脂质代谢紊乱是肿瘤代谢的重要特征之一,一方面脂质代谢为肿瘤细胞的异常增殖供给大量能量;另一方面多种脂质分子是细胞膜、脂质信号分子的重要组分,参与诸多肿瘤相关通路的信号转导,涉及肿瘤(包括HCC)的增殖、侵袭、转移、血管生成等生物学过程[16-17]。目前,笔者暂未发现miR-1180参与调控脂质代谢的相关研究,深入研究miR-1180、脂质代谢、HCC三者间的相关性无疑具有广阔前景。
为进一步揭示miR-1180在HCC中的作用机制,本研究结合靶基因蛋白互作网络及预后分析筛选出6个miR-1180潜在关键靶基因,分别为ETS2、PPARGC1A、ALDH2、SARDH、HMGCS2、ESR1;其中PPARGC1A、ALDH2、HMGCS2、ESR1为脂质代谢调控相关基因。现有研究表明,PPARGC1A、ALDH2、HMGCS2、ESR1等4个关键靶基因与HCC的发生、发生过程密切相关。与本研究结果一致,相关研究已发现PPARGC1A、ALDH2、HMGCS2、ESR1等基因在HCC中均呈现低表达,且与患者不良预后有关;此外,当上调HCC细胞株中上述基因的表达水平则可通过调控相关信号通路(如AMPK信号通路、转录因子SP1等)显著抑制HCC细胞的增殖、迁移或侵袭等生物学过程,提示了PPARGC1A、ALDH2、HMGCS2、ESR1在HCC的发生、发展中发挥抑癌基因的作用[18-22]。目前,ETS2、SARDH与HCC关系的研究鲜有报道。作为MAPK/ERK通路的重要底物之一,ETS2的异常表达与多种肿瘤关系密切,且所起作用具有一定的组织特异性,发挥着癌基因(如喉癌等)或抑癌基因(如肺癌等)的作用[23-24]。SARDH是调控肌氨酸代谢的关键酶之一,其异常表达同样涉及肿瘤的发生与发展。Lim等[25]发现SARDH在HCC组织中较正常肝组织低表达,提示SARDH与HCC的发生、发展紧密相关,但具体作用及机制尚不明确。Khan等[26]发现SARDH在前列腺癌中低表达,当过表达SARDH则可显著抑制癌细胞的增殖能力,发挥抑癌基因的作用。结合本次研究靶基因差异表达分析和预后分析结果,推测ETS2和SARDH在HCC中同样作为抑癌基因而存在。基于上述研究结果及文献复习,我们认为HCC中miR-1180高表达可诱导其关键靶基因(ETS2、PPARGC1A、ALDH2、SARDH、HMGCS2、ESR1等)表达水平的异常上调,并由此促进HCC的发生和发展。
综上所述,本研究基于GEO和TCGA数据库合并系统的生物信息学方法揭示了miR-1180可作为一种促癌基因参与HCC的发生、发展,并拥有作为HCC诊断标志物、预后指标及治疗靶点的潜在应用价值。总之,本次生物信息学分析将有望为日后HCC发病机制的研究以及诊治方法的改善提供新策略、开辟新路径,但miR-1180及其靶基因在HCC中作用及其机制还有待后续实验予以证实及丰富。
[1] Tang A, Hallouch O, Chernyak V, et al. Epidemiology of hepatocellular carcinoma: target population for surveillance and diagnosis[J]. Abdom Radiol (NY), 2018, 43(1):13-25. doi: 10.1007/s00261-017-1209-1.
[2] Rich NE, Yopp AC, Singal AG. Medical Management of Hepatocellular Carcinoma[J]. J Oncol Pract, 2017, 13(6):356-364.doi: 10.1200/JOP.2017.022996.
[3] Catela Ivkovic T, Voss G, Cornella H, et al. microRNAs as cancer therapeutics: A step closer to clinical application[J]. Cancer Lett,2017, 407:113-122. doi: 10.1016/j.canlet.2017.04.007.
[4] 赵新阳, 肖朝文, 郑小林, 等. miR-96的表达对肝细胞癌细胞迁移和侵袭的影响[J]. 中国普通外科杂志, 2017, 26(7):877-882.doi:10.3978/j.issn.1005-6947.2017.07.010.Zhao XY, Xiao CW, Zheng XL, et al. Influence of miR-96 expression on migration and invasion of hepatocellular carcinoma cells[J]. Chinese Journal of General Surgery, 2017, 26(7):877-882.doi:10.3978/j.issn.1005-6947.2017.07.010.
[5] Chen EG, Zhang JS, Xu S, et al. Long non-coding RNA DGCR5 is involved in the regulation of proliferation, migration and invasion of lung cancer by targeting miR-1180[J]. Am J Cancer Res, 2017,7(7):1463-1475.
[6] Gu L, Zhang J, Shi M, et al. The effects of miRNA-1180 on suppression of pancreatic cancer[J]. Am J Transl Res, 2017,9(6):2798-2806.
[7] Wang C, Ge Q, Chen Z, et al. A New Double Stranded RNA Suppresses Bladder Cancer Development by Upregulating p21(Waf1/CIP1) Expression[J]. Biomed Res Int, 2015, 2015:304753. doi: 10.1155/2015/304753.
[8] Varet H, Brillet-Guéguen L, Coppée JY, et al. SARTools: A DESeq2- and EdgeR-Based R Pipeline for Comprehensive Differential Analysis of RNA-Seq Data[J]. PLoS One, 2016,11(6):e0157022. doi: 10.1371/journal.pone.0157022.
[9] Pan C, Wang X, Chen W, et al. Reevaluation of glypican-3 as a prognostic marker in HCC using X-tile software[J]. Med Oncol,2015, 32(1):359. doi: 10.1007/s12032-014-0359-z.
[10] Szklarczyk D, Morris JH, Cook H, et al. The STRING database in 2017: quality-controlled protein-protein association networks, made broadly accessible[J]. Nucleic Acids Res, 2017, 45(D1):D362-368.doi: 10.1093/nar/gkw937.
[11] Brysbaert G, Mauri T, Lensink MF. Comparing protein structures with RINspector automation in Cytoscape[J]. Version 2. F1000Res,2018, 7:563. doi: 10.12688/f1000research.14298.2.
[12] Zhou X, Zhu HQ, Ma CQ, et al. MiR-1180 promoted the proliferation of hepatocellular carcinoma cells by repressing TNIP2 expression[J]. Biomed Pharmacother, 2016, 79:315-320. doi:10.1016/j.biopha.2016.02.025.
[13] Tan G, Wu L, Tan J, et al. MiR-1180 promotes apoptotic resistance to human hepatocellular carcinoma via activation of NF-kappaB signaling pathway[J]. Sci Rep, 2016, 6:22328. doi: 10.1038/srep22328.
[14] Mamdouh S, Khorshed F, Aboushousha T, et al. Evaluation of Mir-224, Mir-215 and Mir-143 as Serum Biomarkers for HCV Associated Hepatocellular Carcinoma[J]. Asian Pac J Cancer Prev,2017, 18(11):3167-3171. doi: 10.22034/APJCP.2017.18.11.3167.
[15] Li P, Dong J, Zhou X, et al. Expression patterns of microRNA-329 and its clinical performance in diagnosis and prognosis of breast cancer[J]. Onco Targets Ther, 2017, 10:5711-5718. doi: 10.2147/OTT.S147974.
[16] Long J, Zhang CJ, Zhu N, et al. Lipid metabolism and carcinogenesis, cancer development[J]. Am J Cancer Res, 2018,8(5):778-791.
[17] Chen X, Gu X, Zhang H. Sidt2 regulates hepatocellular lipid metabolism through autophagy[J]. J Lipid Res, 2018, 59(3):404-415. doi: 10.1194/jlr.M073817.
[18] Liu R, Zhang H, Zhang Y, et al. Peroxisome proliferatoractivated receptor gamma coactivator-1 alpha acts as a tumor suppressor in hepatocellular carcinoma[J]. Tumour Biol, 2017,39(4):1010428317695031. doi: 10.1177/1010428317695031.
[19] Hou G, Chen L, Liu G, et al. Aldehyde dehydrogenase-2 (ALDH2)opposes hepatocellular carcinoma progression by regulating AMP-activated protein kinase signaling in mice[J]. Hepatology, 2017,65(5):1628-1644. doi: 10.1002/hep.29006.
[20] Su SG, Yang M, Zhang MF, et al. miR-107-mediated decrease of HMGCS2 indicates poor outcomes and promotes cell migration in hepatocellular carcinoma[J]. Int J Biochem Cell Biol, 2017, 91(Pt A):53-59. doi:10.1016/j.biocel.2017.08.016.
[21] Tu CC, Kumar VB, Day CH, et al. Estrogen receptor alpha (ESR1)over-expression mediated apoptosis in Hep3B cells by binding with SP1 proteins[J]. J Mol Endocrinol, 2013, 51(1):203-212. doi:10.1530/jme-13-0085.
[22] Hishida M, Nomoto S, Inokawa Y, et al. Estrogen receptor 1 gene as a tumor suppressor gene in hepatocellular carcinoma detected by triple-combination array analysis[J]. Int J Oncol, 2013, 43(1):88-94.doi: 10.3892/ijo.2013.1951.
[23] Kabbout M, Garcia MM, Fujimoto J, et al. ETS2 mediated tumor suppressive function and MET oncogene inhibition in human nonsmall cell lung cancer[J]. Clin Cancer Res, 2013, 19(13):3383-3395.doi: 10.1158/1078-0432.CCR-13-0341.
[24] Liu X, Zhang C, Zhang Z, et al. E26 Transformation-Specific Transcription Factor ETS2 as an Oncogene Promotes the Progression of Hypopharyngeal Cancer[J]. Cancer Biother Radiopharm, 2017, 32(9):327-334. doi: 10.1089/cbr.2017.2296.
[25] Lim SO, Park SJ, Kim W, et al. Proteome analysis of hepatocellular carcinoma[J]. Biochem Biophys Res Commun, 2002, 291(4):1031-1037. doi: 10.1006/bbrc.2002.6547.
[26] Khan AP, Rajendiran TM, Ateeq B, et al. The role of sarcosine metabolism in prostate cancer progression[J]. Neoplasia, 2013,15(5):491-501.