结直肠癌是世界上第三大常见恶性肿瘤。尽管不断努力预防和改进治疗,其预后仍很差,大约35%~40%的局部进展期直肠癌患者最终会发生远处转移并死亡[1],该病病死率与肝脏转移的发生密切相关。结直肠癌的动物模型是研究结直肠癌发生和发展机制实验物质基础。因此,通过动物模型研究结直肠癌肝转移的机制是控制疾病进展的重要研究手段。目前人们已在体外和体内开发了肝转移模型。体外转移模型易于操作,廉价且可重复。这些模型允许操纵转移过程中的每个步骤,但是对整个转移过程的综合分析存在局限性[2-3]。相反, 使用动物的体内转移模型可以更准确地代表转移过程并且可以模拟人体癌症进行相应的基因操作。然而,它们也存在着相关的伦理问题和经济负担[4-6]。目前,已经开发了许多动物模型用于评估肝转移的机制和建立治疗策略[4-9]。这些临床前研究提供了与临床应用相关的证据,并且有助于改善具有肝转移的结肠直肠癌患者的存活。本文就目前用于结直肠癌肝转移的小鼠模型综述如下。
1 小鼠结直肠模型的类型
目前小鼠模型是肿瘤转移研究中应该最广泛的动物模型。小鼠相比其他动物在实验动物学上具有许多优势,小鼠基因组与人类基因组高度同源,在解剖和生理上具有相似性。小鼠的繁殖能力强,生殖周期短,饲养相对简单,体积小,十分有利于进行大规模的实验。小鼠结直肠癌肝转移动物模型类型总结为图1。
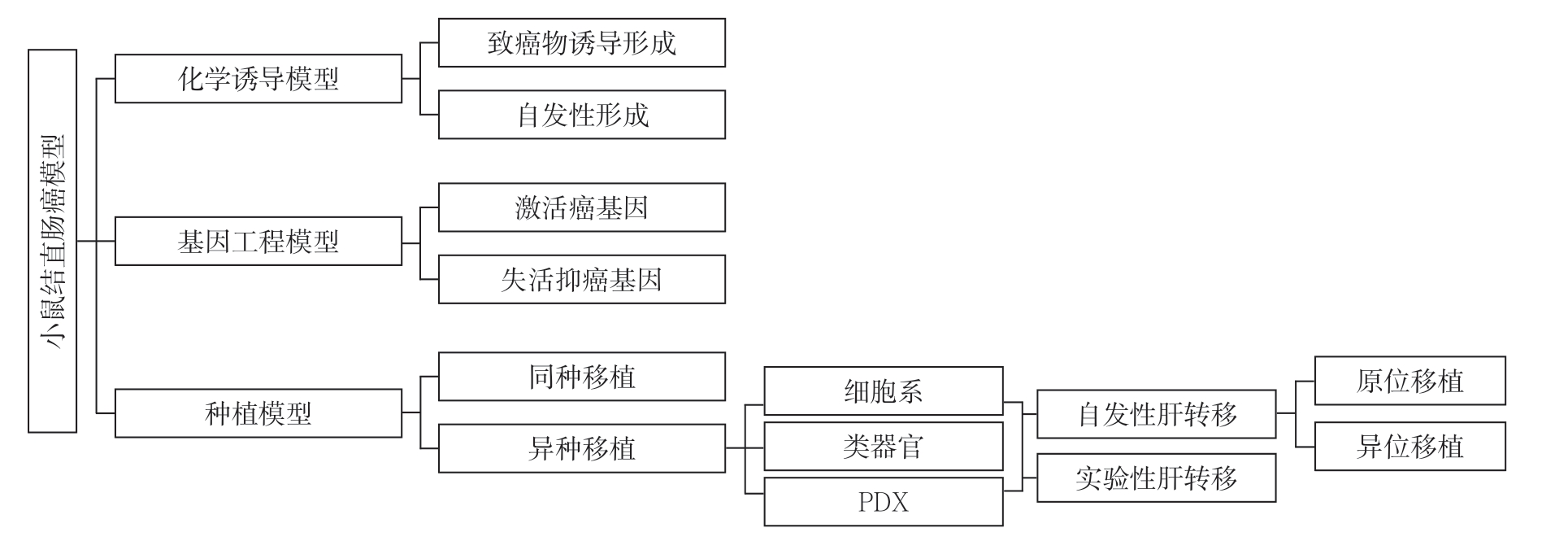
图1 小鼠结直肠癌肝转移模型的类型
Figure 1 Types of mouse models of CRC liver metastasis
1.1 按肿瘤发生的角度分类
从肿瘤发生的角度出发,总的说可以分为三大类型:化学诱导模型、基因工程模型和种植模型。化学诱导模型是指让小鼠暴露在化学致癌物下,使其自发形成癌症或者转移的模型。基因工程模型通过基因组表达的改变而建立,例如癌基因的激活和抑癌基因的失活。种植模型是将来自人(异种)或鼠(同种异体)的移植物植入荷瘤小鼠体内的模型。
1.2 按移植物的种属分类
移植物的种属和是否 保留的肿瘤异质性决定选择什么免疫活性的小鼠,通常有两种选择:在具备免疫活性的小鼠中移植同种异体移植物和在免疫缺陷小鼠中移植异种移植物。在具备免疫活性的小鼠中移植同种异体移植物的方法对于评估肿瘤与其微环境之间的相互作用有较大意义,移植物很少排斥,它们具有经济效益和方便性,然而这些模型不能反映人类肿瘤的遗传异质性。在免疫缺陷小鼠中移植异种移植物的方法尽管存在诸如免疫系统功能缺陷和移植成功率低的局限性,但是随着精准医疗的兴起,是否能够反应人类肿瘤异质性尤为重要,因此异种移植在人类肿瘤研究中也得到了广泛的应用,目前常见的异种移植模型有细胞系、类器官和病人源肿瘤移植(patient-derived tumor xenograft,PDX)[10]。将人结肠直肠癌细胞原位或者异位注射到免疫缺陷小鼠中通常会导致注射部位的肿瘤生长,常用的小鼠品系包括裸鼠(无胸腺)和严重联合免疫缺陷(SCID)小鼠。自2010年初以来,种植类器官的技术迅速发展,类器官是体外三维形成的器官的小型化和简化版,Matano等[11]使用CRISPR/Cas9技术编辑人结直肠癌类器官的APC,TP53,SMAD4,KRAS和/或PIK3CA等基因,然后用以上基因工程化的类器官建立肾被膜移植和实验性肝转移的小鼠模型,结果显示基因工程化的类器官成功在肝脏定植。PDX模型是将手术期间获得患者肿瘤组织的部分并植入免疫缺陷小鼠,一旦肿瘤生长就将其取出植入其他小鼠进行传代,此后重复这一过程,直到获得足够的动物用于实验。PDX模型受到越来越多的关注,它能反映每一位患者的肿瘤异质性,可用于个性化用药指导、药效筛选和生物标志物的开发等。
1.3 按结直肠癌肝转移的建立角度分类
从肝 转移的建立角度出发,可分为自发性转移模型和实验性转移模型。目前,采用结直肠癌细胞系或者肿瘤组织移植到小鼠中建立结直肠癌肝转移动物模型是最为常见的造模方法。自发性肝转移模型就是采用上述方法,在不经过其他干预下经肿瘤自由生长后发生转移的模型;而实验性转移模型则是人为通过静脉、脾脏或其他部位注射肿瘤细胞模拟血道转移的模型,具体见后文。
2 化学诱导性模型、基因工程模型和PDX模型:完整肿瘤微环境的分歧
由 于肿瘤微环境对肿瘤进展具有十分重要的作用,因此在有免疫活性的小鼠中建立结直肠癌肝转移模型很有必要。有免疫活性的动物表现出的健康状态通常更稳定,其完整的免疫系统可用于测试免疫疗法的效果,而且将免疫机制作为癌症的治疗靶点进行研究已成为热点之一。在有免疫活性的小鼠中通过化学致癌物诱导和基因工程技术可以满足在原发部位形成完整肿瘤微环境的特点,但其并不能体现肿瘤异质性。而PDX模型虽然克服了前两者缺乏肿 瘤异质性的缺点,但是免疫系统受损,缺乏免疫细胞的参与,肿瘤微环境不完善。
2.1 化学诱导模型
自发和致癌物诱导的结直肠癌的小鼠模型是最早有相关报道的模型。小鼠自发形成结 直肠癌的发生率为1%[12-13],因此无法对结直肠癌的进展进行有效地实验研究。为了增加结直肠癌的发病率,将小鼠暴露于致癌物下,大多数致癌物质会引起多个器官的恶性肿瘤,但有些会主要诱发结直肠癌。经常使用的致癌物质有二甲肼(dimethylhydrazine,DMH)及其代谢产物氧化偶氮甲烷(azoxymethane,AOM),杂环胺(Heterocyclic amines,HCAs),甲基硝基亚硝基胍(N-Methyl-N-nitro-N-nitrosoguanidine,MNNG)和甲基亚硝基脲(N-methyl-N-nitrosourea,MNU)[14]。研究[15-16]通过皮下和腹膜下重复使用AOM,约30周后出现自发性结肠癌。MNU的直肠内给药导致结肠肿瘤发生率为100%,此外23%~31%的小鼠发生肠系膜淋巴结的转移[17]。
致癌物诱导的结直肠癌模型在基因-环境相互作用和化学预防的研究中具有优势[18],但是缺点是肿瘤发生率低,因此必须使用大量的实验动物。此外,该模型不适合结直肠癌晚期和转移形成机制研究,因为这种模型转移进展非常缓慢且不常发生。
2.2 基因工程模型
通过研究家族性腺瘤性息肉病(FAP)和遗传性非息肉性结肠癌(HNPCC),揭示了结肠癌发生过程中存在基因突变[19]。具有某些基因突变的小鼠显示出与人结肠癌和息肉相似的表型。从局部增生到腺瘤再到浸润性癌最后发生转移,这多阶段的过程需要各种基因的连续突变。因此人们开始使用基因 工程小鼠来研究结直肠癌肝转移。
结直肠癌的基因工程小鼠模型通常使用特定种系或遗传修饰,这样可用于研究遗传性结直肠癌[6]。然而,散发性结直肠癌发生的是体细胞突变,占所有结肠直肠癌约80%,因此需要体细胞工程小鼠模型来研究结直肠癌的发生[20]。结直肠癌的大多数基因工程小鼠模型是根据杂合性丧失时诱导APC突变而产生的,尽管APC突变可导致侵袭性结直肠癌的形成,但尚未在这些模型中证实肝转移[21]。最近,建立了一个相当高的肝转移发展效率的结直肠癌基因工程小鼠模型。该模型是将Adeno-Cre注射到LSL-KRASG12V/APCflox/flox小鼠的结肠中产生,当结肠感染腺病毒诱导APC缺失和致癌KRASG12V激活,然后散发性结肠直肠癌的产生,随后肝转移[22]。
基因工程小鼠模型具有显著的优点,该模型提供有关癌发生过程中特定基因突变的影响,有效地评估肿瘤发生的早期步骤,有助于与结肠癌的发病机制相结合。与种植模型相比,基因工程模型更准确地代表了肿瘤进展的自然过程以及肿瘤细胞与微环境之间的相互作用[5]。此外,这些模型具有完整的免疫系统和特异性的微环境[23]。
但是它们缺点是通常不能完全重现人体肿瘤的遗传复杂性。另外,从实验角度出发,总体而言肝转移很少发生,并且它们的发生是不可预测的[24],即使发生肝转移,也需要等待很长时间[25],因此其潜在局限性在于难以评估治疗反应[26];在研 究某些特定基因的突变可导致胚胎致死,严重的发育缺陷或不育[27];实验的完成需要承担昂贵经济压力且工作量大[28]。
2.3 PDX模型
皮下移植的PDX模型很少发生肝转移,最常用的PDX肝转移模型是患者来源的原位移植模型。研究[29-30]发现,从患者手术获得的组织学上完整的结肠癌组织在10次传代后,随后原位植入裸鼠盲肠的PDX模型能发生肝转移。其他研究也报道了原位PDX模型发生结直肠癌的肝转移[31-33]。
来自细胞系的异种移植虽然具有重复好、易于操作和良好表型的优点,但是它们没有表现出原始 肿瘤的肿瘤异质性或组织病理学和遗传特征[34-36]。相比之下,PDX模型更好地反映了原始肿瘤的特征和遗传多样性。因此,这些模型目前是用于测试药物反应的最佳临床前模型,特别是对于患有难治性癌症的患者。此外,PDX模型可进一步用于个性化治疗的基因组学和药理学研究[31, 35]。
PDX转移模型的主要优点是这些模型可以检测患者对药物的特异性治疗反应并预测疾病过程,提供关于个性化治疗的关键信息[5, 37]。与来自癌细胞系的异种移植模型相比,PDX模型还在细胞和遗传水平上保持原始肿瘤的异质性[34, 31-32]。此外,PDX转移模型提供了跟踪转移的起始和进展的机会[37]。
尽管具有这些优点,但PDX转移模型在转移研究中具有许多缺点。已经表明,人肿瘤基质最初在移植后可以保存,但随着时间的推移会逐渐被鼠基质取代[38]。尽管PDX是个性化医疗的希望,但PDX移植成功率并不高[39],这可能会限制临床效用。另外,肿瘤植入和筛选潜在疗法可能需要6个月或更长时间。最后,临床上通过PDX模型来进行诊疗价格十分昂贵,并且私人信息有时难以得到有效保护。
3 自发性肝转移模型和实验 性肝转移模型:模拟转移途径的分歧
3.1 自发性肝转移模型
自发性肝转移模型是指将癌细胞系或组织植入结肠(原位移植)或皮下层(异位移植),导致在注射部位形成原发性肿瘤,随后自行发生肝转移。异位移植是肿瘤移植最简单的方法。然而,这种模型大多不能发展肝脏转移,因为皮下微环境与结肠完全不同 。相比之下,原位移植模型在组织学,血管分布,基因表达和转移过程方面类似于人体癌症。有学者[40]提出该模型应该满足:首先,原发肿瘤必须定位在结肠;第二,原发性肿瘤必须来自结肠壁的黏膜层;第三,单个完整的肿瘤应植入黏膜层(以保持肿瘤细胞与基质细胞和细胞外基质的相互作用);第四,应在肝、肺和淋巴结中检测到转移性病变。因此,自发性肝转移模型主要选用原位移植模型。在该模型中,可以通过开放手术、灌肠、显微注射和经肛门低剂量电凝等手段将结肠癌细胞系或人肿瘤组织移植到免疫受损小鼠的结肠壁中[41]。
为确保肝转移的有效进展,体内选择高转移性肿瘤细胞是一项重要的策略。通过连续传代可以增加移植肿瘤的转移潜力。在这种方法中,致瘤样本扩增后重新植入盲肠重复几代,以获得更高的致瘤性和转移能力。这种具有高度转移潜能的原位移植模型已被证明可有效进展为肝转移。然而,这种选择过程和随后的体外细胞培养导致肿瘤细胞在二维环境中更适应生长而不是在正常的三维环境中。
原位自发性肝转移模型的主要优点是转移遵循自然过程并利用类似于人体的机制。因此,该模型适用于预测人体肿瘤中的药物反应。该模型代表着从原发部位形成到进展为肝转移的整个转移过程。它能够研究转移级联中的所有步骤,可以模仿人体肿瘤微环境,并在肿瘤细胞中提供真实水平的异质性。此外,在自发转移模型中分离的高转移性肿瘤细胞所需的传代次数少于实验性转移模型所需的传代次数 。
该模型的缺点是从结肠到肝脏的自发转移为低预测性和差重复性。由于异种移植物中缺乏功能性免疫系统,因此该模型难以评估免疫系统对转移进展的作用。此外,发生肝转移需要很长时间,有时需要切除原发肿瘤以减轻肿瘤负荷从而使转移发生。此外,盲肠注射在技术上存在困难,并且存在肿瘤细胞渗漏或管腔内溢出的风险。
3.2 实验性肝转移模型
实验性肝转移模型是指实验性地诱导肝转移。实验性转移模型发生在不同器官中,取决于注射的部位和注射细胞的向性。在实验性肝转移模型中,是将结肠癌细胞直接注射到脾脏或门静脉中。肿瘤细胞在不经历原发性肿瘤生长和血管内渗出等步骤的情况下到达肝脏微循环,然后因大小或特定的因素而滞留。这些肿瘤细胞经历细胞外渗并进入肝实质,或者它们可能直接在脉管系统内生长。部分注射细胞最终形成肝转移瘤。为了提高肝转移的效率,先前描述的在自发模型中连续传代方法在实验模型中是必要的。有报道称,当人体结肠癌细胞注入免疫受损小鼠的脾脏时,高度恶性的细胞会发生肝脏转移[3, 7, 9, 42-43]。
实验性转移模型为转移研究提供了许多优势。肿瘤细胞直接注入体循环,因此转移发生需要的时间很短。此外,这些模型具有高度可重复性并且始终导致转移形成。与自发转移模型相比,这些模型中的转移形成更有效。使用这些模型能够控制引入循环中的肿瘤细胞的数量和类型,这对于实现实验终点十分重要。此外,转移器官可以靶向特定部位[3, 42, 44-45]。
实验性转移模型的主要缺点是这些模型不能代表整个转移过程。 它们仅代表转移级联的晚期,因为它们绕过早期阶段,包括原发性肿瘤生长和肿瘤细胞的内渗。应通过连续传代分离肿瘤细胞,以增强其组织特异性能力。转移遵循人工途径,而不是其自然过程[3, 9, 42]。
4 理想的小鼠 结直肠癌癌肝转移模型
适合对肿瘤转移研究的理想动物模型应该具备一些条件和因素,例如:不管是原发性肿瘤还是转移性肿瘤都应该可预测和重复,能够得到广泛应用;保持肿瘤的异质性和微环境;在动物体内能重建完整的肿瘤转移过程;从模型的应用价值出发,应该可以用来做肿瘤患者的药效学评价或者新治疗手段的测试。但是目前很难同时满足所有条件,应当根据具体实验需要选择恰当的模型。例如侧重研究肿瘤微环境和免疫相关机制时,在免疫缺陷动物中移植异种细胞则不适合;侧重研究肿瘤的转移途径时,则应该在结直肠原位形成肿瘤,让其自发形成肝转移,而不是在肝脏直接形成肿瘤。
5 小结与展望
各小鼠模型各自有优缺点详见表1。建立结直肠癌肝转移的小鼠模型有助于对转移过程和机制的了解。理想的肝转移模型应该建立可预测和可重复的条件上。此外,未来原发性肿瘤模型应能同时拟合结直肠癌肿瘤(表观)遗传学、病理学、解剖学特性,其结直肠癌肝转移模型具备短时间自发发生肝转移的能力,以此来有效地代表人体肿瘤微环境,并可用于验证多种治疗癌症患者。虽然目前应用于肝转移的理想模型方面仍存在局限性,但幸运的是,基因组编辑技术的发展,如CRISPR/Cas系统,已经为肝转移模型的发展提供了新的手段,关于肿瘤发生和转移机制的表观遗传学研究也正是目前基础医学研究的热点之一,这些进展未来必将会为结直肠癌肝转移模型带来革新。总之,此类动物模型的目标是通过对其潜在转移机制研究为患有难治性结肠直肠癌的患者设计有效的治疗方法。
表1 小鼠肝转移模型的优缺点
Table 1 Advantages and disadvantages of models of different mouse models of CRC liver metastasis
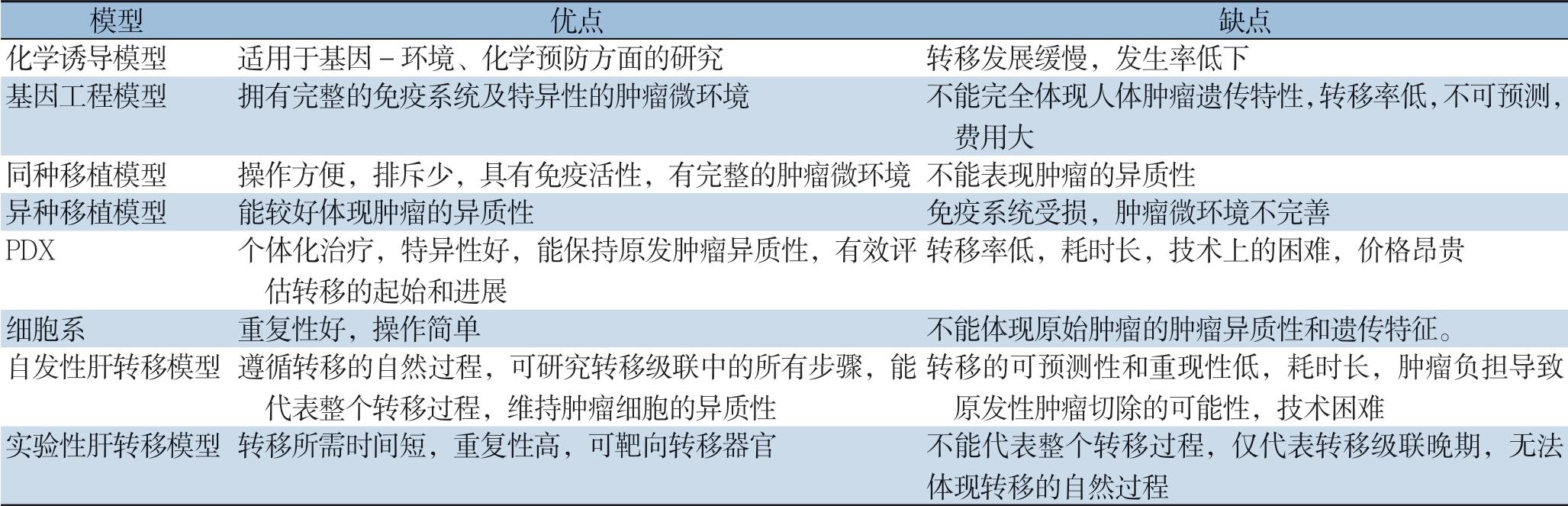
注:部分造模手段存在交叉,可根据实际需求自行搭配
Note: Model selection is f l exible because of the overlaps among some model creation methods
模型 优点 缺点化学诱导模型 适用于基因-环境、化学预防方面的研究 转移发展缓慢,发生率低下基因工程模型 拥有完整的免疫系统及特异性的肿瘤微环境 不能完全体现人体肿瘤遗传特性,转移率低,不可预测,费用大同种移植模型 操作方便,排斥少,具有免疫活性,有完整的肿瘤微环境 不能表现肿瘤的异质性异种移植模型 能较好体现肿瘤的异质性 免疫系统受损,肿瘤微环境不完善PDX 个体化治疗,特异性好,能保持原发肿瘤异质性,有效评估转移的起始和进展转移率低,耗时长,技术上的困难,价格昂贵细胞系 重复性好,操作简单 不能体现原始肿瘤的肿瘤异质性和遗传特征。自发性肝转移模型 遵循转移的自然过程,可研究转移级联中的所有步骤,能代表整个转移过程,维持肿瘤细胞的异质性转移的可预测性和重现性低,耗时长,肿瘤负担导致原发性肿瘤切除的可能性,技术困难实验性肝转移模型 转移所需时间短,重复性高,可靶向转移器官 不能代表整个转移过程,仅代表转移级联晚期,无法体现转移的自然过程
[1]Huang MY, Wu CH, Huang CM, et al. DPYD, TYMS, TYMP, TK1, and TK2 genetic expressions as response markers in locally advanced rectal cancer patients treated with f l uoropyrimidine-based chemoradiotherapy[J]. Biomed Res Int, 2013, 2013:931028. doi: 10.1155/2013/931028.
[2]van Marion DM, Domanska UM, Timmer-Bosscha H, et al. Studying cancer metastasis: Existing models, challenges and future perspectives[J]. Crit Rev Oncol Hematol, 2016, 97:107-117. doi: 10.1016/j.critrevonc.2015.08.009.
[3]Saxena M, Christofori G. Rebuilding cancer metastasis in the mouse[J]. Mol Oncol, 2013, 7(2):283-296. doi: 10.1016/j.molonc.2013.02.009.
[4]Smakman N, Martens A, Kranenburg O, et al. Validation of bioluminescence imaging of colorectal liver metastases in the mouse [J]. J Surg Res, 2004, 122(2):225-230. doi: 10.1016/j.jss.2004.05.021.
[5]Richmond A, Su Y. Mouse xenograft models vs GEM models for human cancer therapeutics [J]. Dis Model Mech, 2008, 1(2/3):78-82. doi: 10.1242/dmm.000976.
[6]Taketo MM, Edelmann W. Mouse models of colon cancer [J]. Gastroenterology, 2009, 136(3):780-798.
[7]Goddard ET, Fischer J, Schedin P. A Portal Vein Injection Model to Study Liver Metastasis of Breast Cancer[J]. J Vis Exp, 2016, (118). doi: 10.3791/54903.
[8]Lee WY, Hong HK, Ham SK, et al. Comparison of colorectal cancer in differentially established liver metastasis models[J]. Anticancer Res, 2014, 34(7):3321-3328.
[9]Zhang Y, Davis C, Shah S, et al. IL-33 promotes growth and liver metastasis of colorectal cancer in mice by remodeling the tumor microenvironment and inducing angiogenesis [J]. Mol Carcinog, 2017, 56(1):272-287. doi: 10.1002/mc.22491.
[10]欧阳国庆, 潘光栋, 吴永融, 等. 小鼠结直肠癌肝转移原位瘤模型[J]. 世界华人消化杂志, 2018, 26(8):512-517. doi: 10.11569/wcjd.v26.i8.512.Ouyang GQ, Pan GD, Wu YR, et al. Orthotopic mouse models of colorectal cancer liver metastases[J]. World Chinese Journal of Digestology, 2018, 26(8):512-517. doi: 10.11569/wcjd.v26.i8.512.
[11]Matano M, Date S, Shimokawa M, et al. Modeling colorectal cancer using CRISPR-Cas9-mediated engineering of human intestinal organoids[J]. Nat Med, 2015, 21(3):256-262. doi: 10.1038/nm.3802.
[12]Anisimov VN, Zabezhinski MA, Rossolini G, et al. Long-live euthymic BALB/c-nu mice. II: spontaneous tumors and other pathologies [J]. Mech Ageing Dev, 2001, 122(5):477-489.
[13]Sødring M, Gunnes G, Paulsen JE. Spontaneous initiation, promotion and progression of colorectal cancer in the novel A/J Min/+ mouse[J]. Int J Cancer, 2016, 138(8):1936-1946. doi: 10.1002/ijc.29928.
[14]Oh BY, Hong HK, Lee WY, et al. Animal models of colorectal cancer with liver metastasis[J]. Cancer Lett, 2017, 387:114-120. doi: 10.1016/j.canlet.2016.01.048.
[15]Grill JI, Neumann J, Ofner A, et al. Dro1/Ccdc80 inactivation promotes AOM/DSS-induced colorectal carcinogenesis and aggravates colitis by DSS in mice[J]. Carcinogenesis, 2018, 39(9):1176-1184. doi: 10.1093/carcin/bgy077.
[16]Du Q, Wang Q, Fan H, et al. Dietary cholesterol promotes AOM-induced colorectal cancer through activating the NLRP3 inflammasome[J]. Biochem Pharmacol, 2016, 105:42-54. doi: 10.1016/j.bcp.2016.02.017.
[17]Iliemene UD, Atawodi SE. Preventive potential of dietary inclusion of Brachystegia eurycoma (Harms) seeds on N-methyl-N-nitrosourea-induced colon carcinogenesis in Wistar rats[J]. J Ethnopharmacol, 2019, 3:111858. doi: 10.1016/j.jep.2019.111858. [Epub ahead of print]
[18]Perše M, Cerar A. Morphological and molecular alterations in 1,2 dimethylhydrazine and azoxymethane induced colon carcinogenesis in rats[J]. J Biomed Biotechnol, 2011, 2011:473964. doi: 10.1155/2011/473964.
[19]Ma H, Brosens LAA, Offerhaus GJA, et al. Pathology and genetics of hereditary colorectal cancer[J]. Pathology, 2018, 50(1):49-59. doi: 10.1016/j.pathol.2017.09.004.
[20]Hung KE, Maricevich MA, Richard LG, et al. Development of a mouse model for sporadic and metastatic colon tumors and its use in assessing drug treatment[J]. Proc Natl Acad Sci U S A, 2010, 107(4):1565-1570. doi: 10.1073/pnas.0908682107.
[21]Rampetsreiter P, Casanova E, Eferl R. Genetically modified mouse models of cancer invasion and metastasis[J]. Drug Discov Today Dis Models, 2011, 8(2/3):67-74. doi: 10.1016/j.ddmod.2011.05.003.
[22]Betzler AM, Kochall S, Blickensdörfer L, et al. A Genetically Engineered Mouse Model of Sporadic Colorectal Cancer[J]. J Vis Exp, 2017, (125). doi: 10.3791/55952.
[23]Firestone B. The challenge of selecting the 'right' in vivo oncology pharmacology model[J]. Curr Opin Pharmacol, 2010, 10(4):391-396. doi: 10.1016/j.coph.2010.06.012
[24]Heijstek MW, Kranenburg O, Borel Rinkes IH. Mouse models of colorectal cancer and liver metastases[J]. Dig Surg, 2005, 22(1/2):16-25. doi: 10.1159/000085342.
[25]Khanna C, Hunter K. Modeling metastasis in vivo [J]. Carcinogenesis, 2005, 26(3):513-523. doi: 10.1093/carcin/bgh261.
[26]Mirantes, Eritja N, Dosil MA, et al. An inducible knockout mouse to model the cell-autonomous role of PTEN in initiating endometrial, prostate and thyroid neoplasias[J]. Dis Model Mech, 2013, 6(3):710-720. doi: 10.1242/dmm.011445.
[27]Ward JM, Elmore SA, Foley JF. Pathology methods for the evaluation of embryonic and perinatal developmental defects and lethality in genetically engineered mice[J]. Vet Pathol, 2012, 49(1):71-84. doi: 10.1177/0300985811429811.
[28]DranoffG. Experimental mouse tumour models: what can be learnt about human cancer immunology?[J]. Nat Rev Immunol, 2011, 12(1):61-66. doi: 10.1038/nri3129.
[29]Kageyama K, Ohara M, Saito K, et al. Establishment of an orthotopic patient-derived xenograft mouse model using uveal melanoma hepatic metastasis[J]. J Transl Med, 2017, 15(1):145. doi: 10.1186/s12967-017-1247-z.
[30]Brown KM, Xue A, Julovi SM, et al. Using patient-derived xenograft models of colorectal liver metastases to predict chemosensitivity[J]. J Surg Res, 2018, 227:158-167. doi: 10.1016/j.jss.2018.02.018.
[31]Puig I, Chicote I, Tenbaum SP, et al. A personalized preclinical model to evaluate the metastatic potential of patient-derived colon cancer initiating cells[J]. Clin Cancer Res, 2013, 19(24):6787-6801. doi: 10.1158/1078-0432.CCR-12-1740.
[32]Oh BY, Lee WY, Jung S, et al. Correlation between tumor engraftment in patient-derived xenograft models and clinical outcomes in colorectal cancer patients [J]. Oncotarget, 2015, 6(18):16059-16068. doi: 10.18632/oncotarget.3863.
[33]Julien S, Merino-Trigo A, Lacroix L, et al. Characterization of a large panel of patient-derived tumor xenografts representing the clinical heterogeneity of human colorectal cancer [J]. Clin Cancer Res, 2012, 18(19):5314-5328. doi: 10.1158/1078-0432.CCR-12-0372.
[34]Cho YB, Hong HK, Choi YL, et al. Colorectal cancer patientderived xenografted tumors maintain characteristic features of the original tumors[J]. J Surg Res, 2014, 187(2):502-509. doi: 10.1016/j.jss.2013.11.010.
[35]Marangoni E, Vincent-Salomon A, Auger N, et al. A new model of patient tumor-derived breast cancer xenografts for preclinical assays[J]. Clin Cancer Res, 2007, 13(13):3989-3998. doi: 10.1158/1078-0432.CCR-07-0078.
[36]John T, Kohler D, Pintilie M, et al. The ability to form primary tumor xenografts is predictive of increased risk of disease recurrence in early-stage non-small cell lung cancer [J]. Clin Cancer Res, 2011, 17(1):134-141. doi: 10.1158/1078-0432.CCR-10-2224.
[37]Siolas D, Hannon GJ. Patient-derived tumor xenografts: transforming clinical samples into mouse models[J]. Cancer Res, 2013, 73(17):5315-5319. doi: 10.1158/0008-5472.CAN-13-1069.
[38]Rosfjord E, Lucas J, Li G, et al. Advances in patient-derived tumor xenografts: from target identification to predicting clinical response rates in oncology[J]. Biochem Pharmacol, 2014, 91(2):135-143. doi: 10.1016/j.bcp.2014.06.008.
[39]Aparicio S, Hidalgo M, Kung AL. Examining the utility of patientderived xenograft mouse models[J]. Nat Rev Cancer, 2015, 15(5):311-316. doi: 10.1038/nrc3944.
[40]Leake I. Colorectal cancer. Understanding the routes of metastasis in colorectal cancer, Nat Rev Gastroenterol Hepatol[J]. Nat Rev Gastroenterol Hepatol, 2014, 11(5):270. doi: 10.1038/nrgastro.2014.60.
[41]Mittal VK, Bhullar JS, Jayant K. Animal models of human colorectal cancer: Current status, uses and limitations[J]. World J Gastroenterol, 2015, 21(41):11854-11861. doi: 10.3748/wjg.v21.i41.11854..
[42]Huang X, Zou Y, Lian L, et al. Changes of T cells and cytokines TGF-β1 and IL-10 in mice during liver metastasis of colon carcinoma: implications for liver anti-tumor immunity [J]. J Gastrointest Surg, 2013, 17(7):1283-1291. doi: 10.1007/s11605-013-2194-5.
[43]Qi J, Yu Y, Akilli Öztürk Ö, et al. New Wnt/β-catenin target genes promote experimental metastasis and migration of colorectal cancer cells through different signals [J]. Gut, 2016, 65(10):1690-1701. doi: 10.1136/gutjnl-2014-307900.
[44]Francia G, Cruz-Munoz W, Man S, et al. Mouse models of advanced spontaneous metastasis for experimental therapeutics [J]. Nat Rev Cancer, 2011, 11(2):135-141. doi: 10.1038/nrc3001.
[45]Kang Y. Analysis of cancer stem cell metastasis in xenograft animal models[J]. Methods Mol Biol, 2009, 568:7-19. doi: 10.1007/978-1-59745-280-9_2.
