近年来,肿瘤已成为全世界人类死亡的主要原因之一。据统计,2018年全球共有1 810万例新发癌症病例和960万例癌症死亡病例,其中肺癌最常被诊断出的癌症(占总病例的11.6%),也是导致癌症死亡的主要原因(占总癌症死亡人数的18.4%)[1]。手术切除仍被认为是大多数早期癌症的主要疗法,而近几十年来,虽然诊断和治疗技术取得了令人鼓舞的进展,然而不幸的是,晚期癌症仍然预后不良。一些临床指标,如组织学分级、病理分型、淋巴结转移和其他临床病理指标,常被用来评估肿瘤的预后。但是,这些指标均不能视为独立的预测指标。因此,继续寻找新的标志物可能为肿瘤的预后预测提供帮助。
人类肝激酶B1(liver kinase B1,LKB1)或丝氨酸-苏氨酸激酶11(serine/threonine kinase 11,STK11)基因位于19号染色体上[2]并在多种人体组织中广泛表达,其编码的LKB1蛋白是一种丝氨酸/苏氨酸激酶,这种激酶可以调节多种细胞的生理和病理过程[3]。LKB1可以直接促进AMPK以调节mTOR通路[4]。LKB1的突变可以导致其失活进而影响癌症相关的代谢,分化[5-7]以及细胞外基质重塑[8],从而促进癌变,局部进展和转移[9]。LKB1还能够对细胞极性和细胞完整性产生影响[10].细胞极性的破坏表明上皮-间质转化(EMT)和癌症进展[11-12]。因此,LKB1目前被认为是许多癌症类型中的关键癌症抑制基因[13]。最新一项研究表明盐诱导激酶(salt-inducible kinases,SIK)对于限制肿瘤发展至关重要,而LKB1和SIK缺陷型肿瘤之间的组织学和基因表达高度相似,这表明LKB1和SIK构成肿瘤抑制轴从而抑制肿瘤进展[14-15]。另一项研究发现LKB1可以抑制调节性T细胞(Treg)和Th17细胞,进而调节树突状细胞的代谢,充当其关键信号枢纽,增强正常的免疫稳态和抗肿瘤能力[16]。LKB1抑制肿瘤进展的机制极为复杂,需要更进一步的研究。
LKB1的低表达与大多数肿瘤的预后不良有关,但也有少数肿瘤类型文章观点相互矛盾[17-18]。在某些特定条件下,LKB1可能是癌症的起始因子[10]。笔者在先前的Meta分析以及最近3年发表了几项新研究中发现了一些有争议的问题[19],因此笔者进行了最新的Meta分析,以评估LKB1表达在癌症患者中的预后价值。
1 资料与方法
按照系统评价和Meta分析的最佳报告声明(PRISMA)的指导进行研究的系统评价和Meta分析[20]。因为Meta分析收集了以前的研究结果并进行了分析,所以未申请伦理审批。
1.1 纳入与排除标准
1.1.1 纳入标准 ⑴ 回顾性队列研究/病例对照研究;⑵ 研究与LKB1表达和癌症预后的关系有关;⑶ 通过免疫组织化学、蛋白质印迹、基因测序、分子分析等方法测定LKB1蛋白的表达水平或基因拷贝数;⑷ 文献中包含足够有效的数据来评估生存结果,包括危险比(HR)、相对比(RR)或优势比(OR)以及相应的整体生存率(OS)95%置信区间(CI),无病生存率(DFS)、无进展生存率(PFS)、无复发生存率/(RFS)等;⑸ 英文文献。
1.1.2 排除标准 ⑴ 重复性文章;⑵ 阅读标题和摘要后无关的文章;⑶ 会议摘要、评论、病例报告、信函、机制研究和动物模型研究;⑷ 缺乏足够信息和数据的研究;⑸ 同一研究中心或同一作者重复发表的文献。
1.2 检索策略
在PubMed、Embase和Cochrane Library数据库中对2 0 1 8年1 2月1日前发表的相关文章进行了系统和全面的搜索。英文检索式包括:LKB1;STK11; Liver Kinase b1(all fields); Neoplasia; Neoplasias;Neoplasm;Tumors;Tumor;Cancer; Cancers;Malignant Neoplasms;Malignant Neoplasm;Neoplasm;Malignant;Neoplasms;Malignant;Malignancy;Malignancies(all fields);Prognosis;Prognostic;Survival;Overall Survival(all fields)。PubMed、Embase和Cochrane Library数据库由2名作者独立搜索。为了避免由于电子筛选方法而造成的遗漏,还对随附文献的参考文献进行了筛选。
1.3 资料提取及文献质量评价
根据上述纳入标准,由3位研究人员客观地提取和纯化符合条件的信息:第一作者;出版年份;地区;癌症类型;男/女;肿瘤分期;中位年龄(范围);结局变量的HR(95% CI);检测方式;HR来源;NOS评分;影响因素;研究类型;是否为多元分析。为避免不必要的偏差,上述所有数据都是由3位研究人员独立提取。
如果在某些文章中同时描述了单变量和多变量分析,则选择多变量分析以保证本研究的准确性。NOS(纽卡斯尔-渥太华质量评估量表)评分用于评估所选研究的质量[21],如果一篇文章的得分为5分以上,则应将其包括在我们的研究中,且≥6分属于高质量。研究质量评估由2名研究人员实施。
1.4 统计学处理
使用STATA/SE 12.0版本(Stata Corporation,College Station,TX,USA)软件来分析和计算统计数据。合并的HR和相应的95% CI用于描述LKB1表达的预后价值;统计异质性通过Cochran Q检验和I2检验进行评估。该软件可用于计算I2和P值,如果I2<50%或P>0.05,则表明纳入研究中的异质性较低,应选择固定效应模型,否则,应该选择一个随机效应模型。异质性的来源可以通过亚组分析和根据相似变量的Meta回归来确定。执行敏感性分析以评估组合结果的稳健性和可靠性。由于在所研究的所有人群和亚组中,癌症的预后指标差异不大,HR、OR和RR之间的差异可以忽略不计,因此,OR和RR均被视为HR[22]。使用Begg的漏斗图和Egger检验来评估潜在的发布偏见。当P<0.05时,表示有统计学意义。
2 结 果
2.1 纳入研究的过程及研究的特点
根据检索策略,在Pubmed、Embase和Cochrane Library数据库中共检索到1 630条记录。删除491篇重复的文献后,通过筛选标题和摘要将1 072篇无关的记录排除在外(图1)。查阅全文之后再次排除44条记录,包括缺乏必要的数据(n=23)、会议摘要(n=10)、审阅(n=3)、病例报告(n=4)或者非英语文献(n=4)。最后,剩余23条记录被保留并纳入Meta分析中[17-18,23-43]。共有4 357例来自不同国家(中国,法国,美国和韩国)的病例被纳入了这项研究,以阐明LKB1表达水平与各种肿瘤患者预后之间的关系。这些病例包含了多种肿瘤患者,包括肝细胞癌、胃癌、胰腺神经内分泌肿瘤、皮肤恶性黑色素瘤、神经胶质瘤,喉癌、非小细胞肺癌、胰腺导管腺癌、肺腺癌、肝内胆管癌、前列腺癌、结直肠癌或乳腺癌。在这项Meta分析中,22项研究报告了OS,12项研究报告了DFS/PFS/RFS。表1总结了与合格研究中纳入患者的主要临床病理特征。
2.2 质量评估
通过NOS评分法对纳入的23项研究进行了评估,其得分在5~8分之间,中位数为6.65分(表1)。根据NOS评分,得分为6分以上的文献属于高质量文献。因此,本Meta分析可被视为有效的方法。
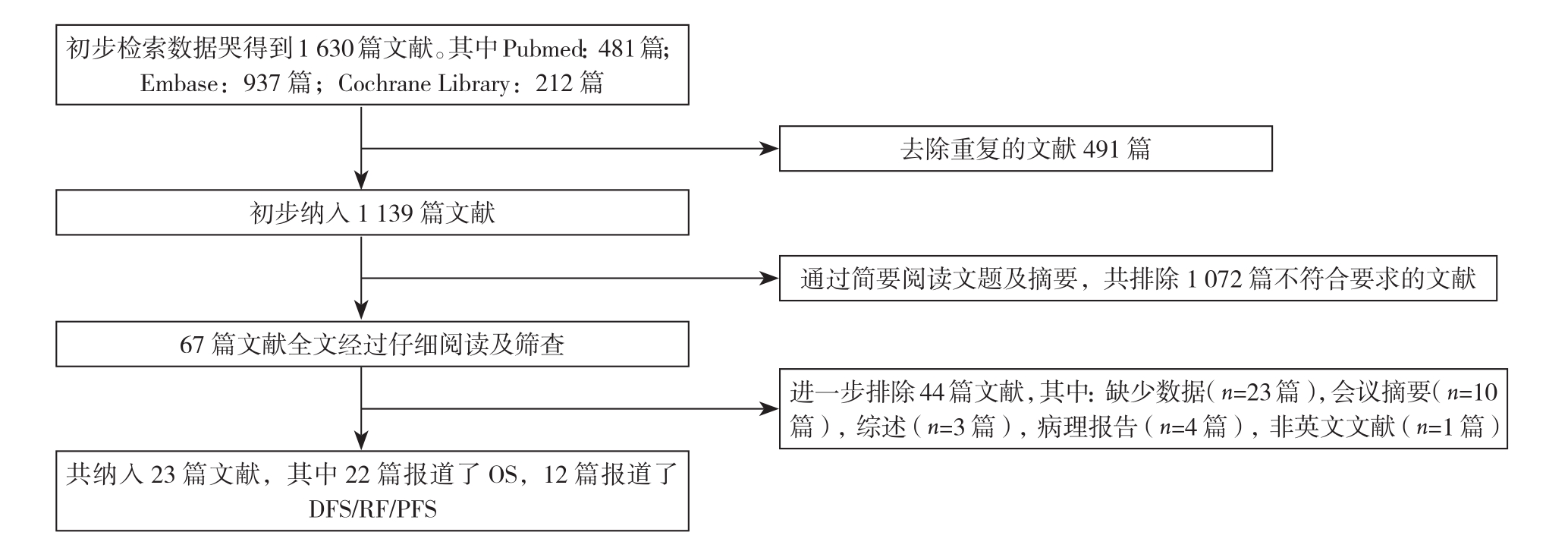
图1 文献纳入流程图
Figure 1 The literature screening process
表1 纳入研究的主要特征
Table 1 Main characteristics of the included studies
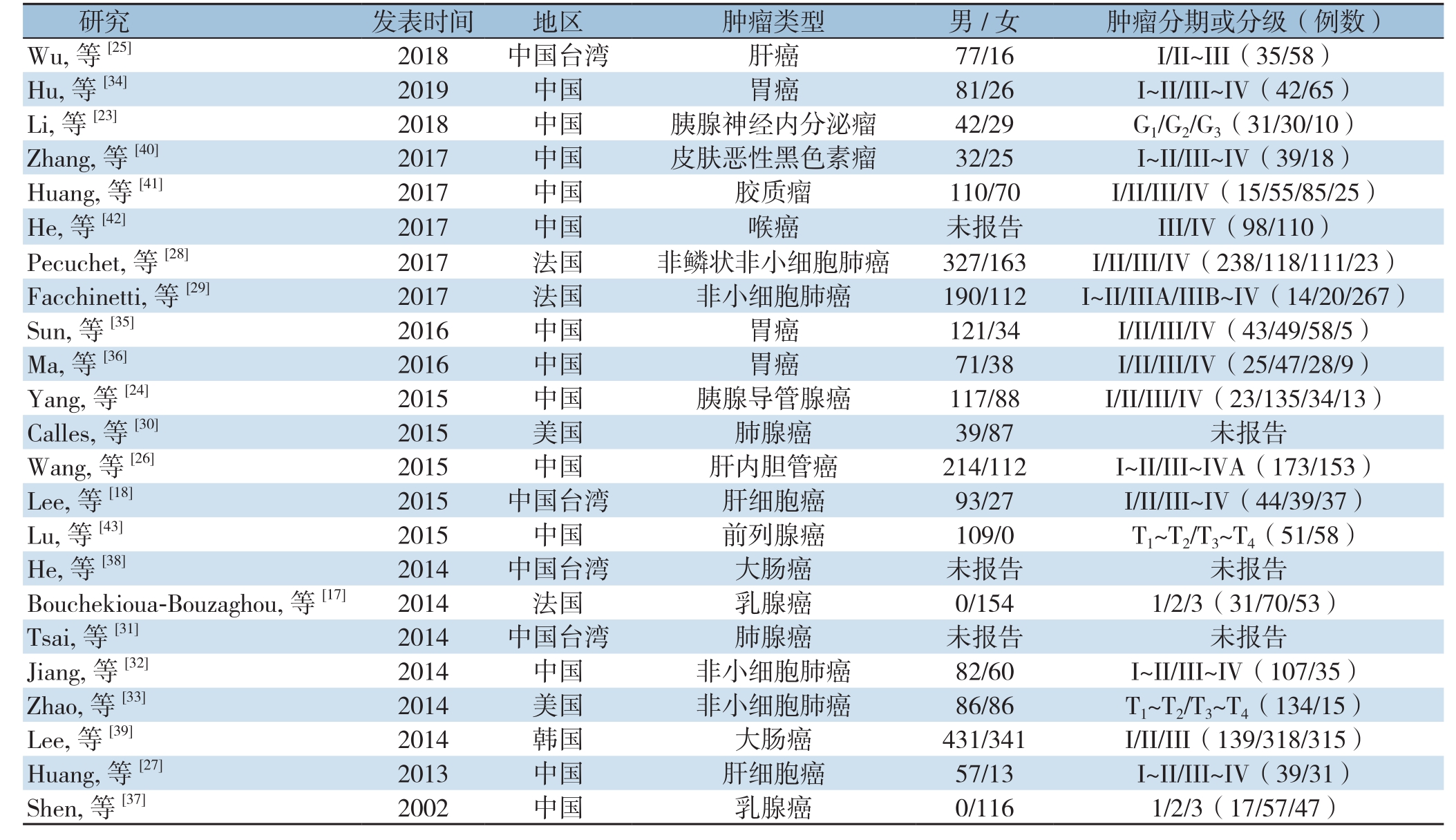
images/BZ_111_211_2088_2267_2137.pngimages/BZ_111_211_2186_2267_3225.pngW u, 等[25] 2018 中国台湾 肝癌 77/16 I/II~III(35/58)Shen, 等[37] 2002 中国 乳腺癌 0/116 1/2/3(17/57/47)
表1 纳入研究的主要特征(续)
Table 1 Main characteristics of the included studies (continue)
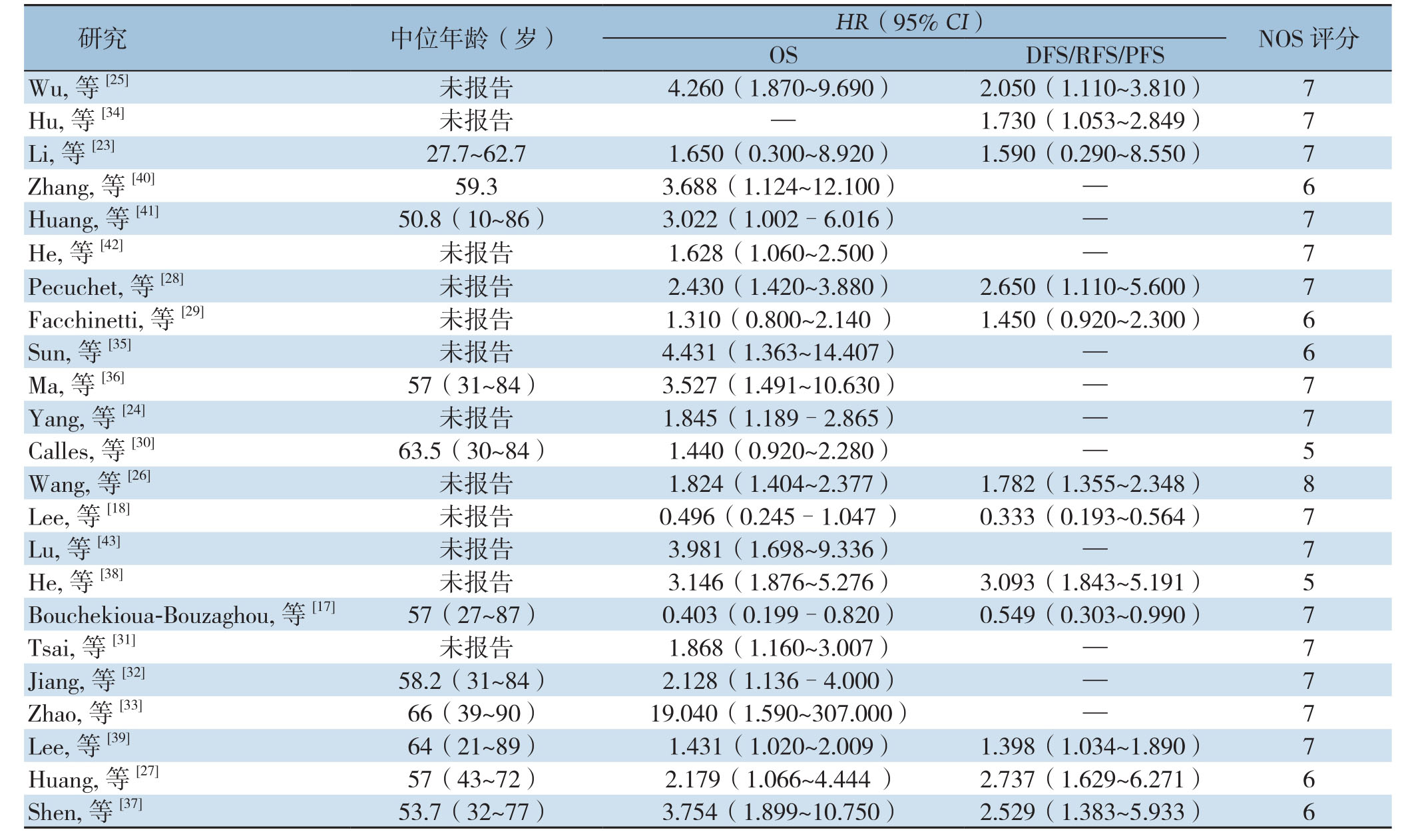
images/BZ_112_211_462_2266_1697.png
2.3 OS和DFS/PFS/RFS
在这项Meta分析中,采用了生存结局变量作为预后指标,即OS、DFS、PFS、RFS;由于P F S和R F S作为结局变量的研究数量有限,将DFS,RFS和PFS结合在一起作为DFS/RFS/P F S来评估预后。鉴于O S观察到统计异质性(I2=65.2%,P<0.001),因此采取了随机效应模型的方法来计算总HR。结果表明低表达的LKB1与肿瘤不良OS之间的明显有关(合并HR=1.94,95% CI=1.55~2.43,P<0.001)。对于DFS/PFS/RFS为结局变量的研究中心同样存在明显的异质性(I2=81.3%,P<0.001),因此也选择了随机效应模型,结果显示,LKB1低表达与不良DFS/PFS/RFS之间明显有关(HR=1.53,95% CI=1.08~2.17,P=0.017)(图2)。综上所述,低表达的LKB1引起肿瘤患者的不良预后。
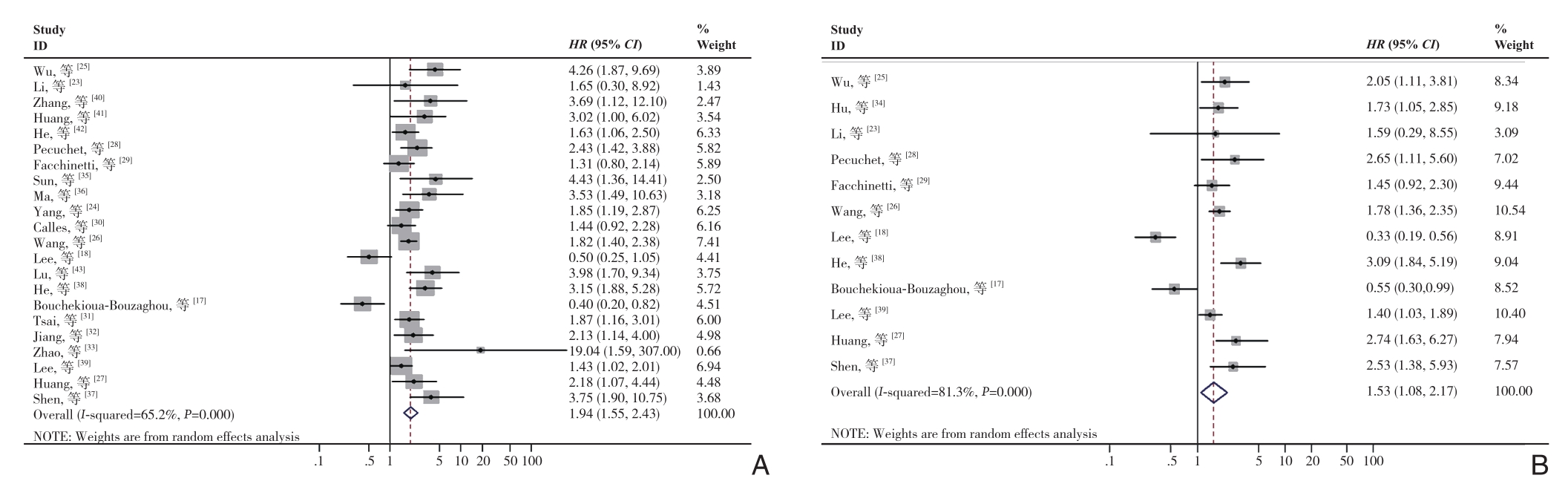
图2 各种肿瘤中LKB1低表达的HR森林图 A:OS的HR;B:DFS/RFS/PFS的HR
Figure 2 Forest plots of HR for low LKB1 expression in various cancers A: HR of OS; B: HR of DFS/RF/PFS
2.4 亚组分析和Meta回归分析
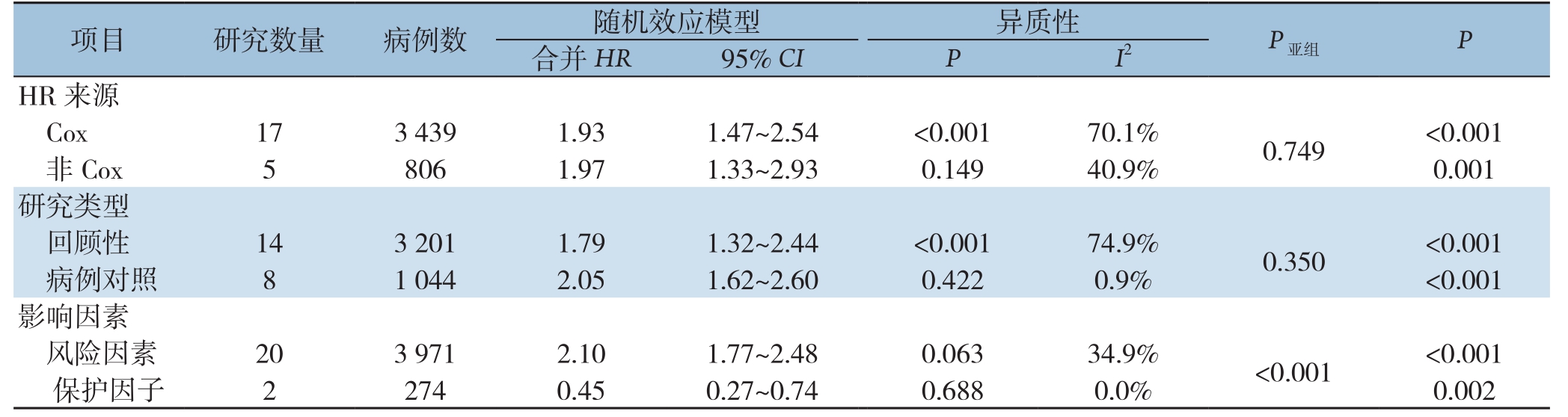
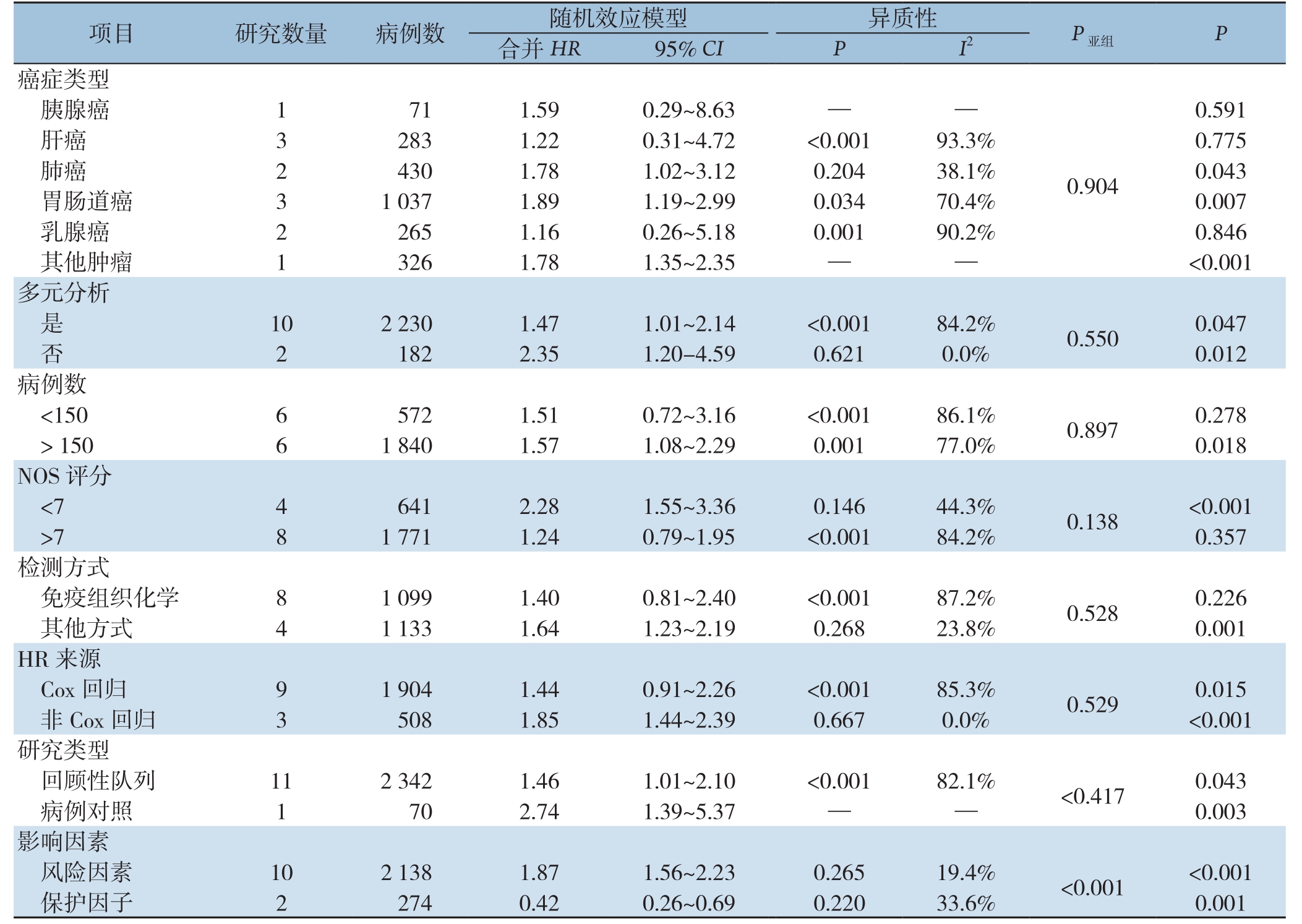
由于所纳入的研究中明显存在统计学异质性,因此进行了亚组分析寻找异质性的来源,主要分析了以下几个混杂因素的影响:癌症类型、NOS评分、研究类型和影响因素。由于报告不足和临界值变化,未对其他特征进行分析以探讨异质性的来源。对于OS(图3),肝癌(HR=1.65,95% CI=0.48~5.66,P=0.428)和乳腺癌(HR=1.21,95% CI=0.14~10.80,P=0.863)在LKB1低表达与不良预后之间没有显示出明确的关系。对于DFS/RFS/PFS(图4),NOS≥7(HR=1.25、95% CI=0.55~2.83,P=0.357)、免疫组化(HR=1.40,95% CI=0.81~2.40,P=0.226)、RFS(HR=1.28,95% CI=0.32~5.14,P=0.727),DFS(HR=1.52,95% CI=0.97~2.38,P=0.066)、肝癌(HR=1.22,95% CI=0.31~4.72,P=0.775)、胰腺癌(HR=1.59、95% CI=0.29~8.63,P=0.591)和乳腺癌(HR=1.16,95% CI=0.26~5.18,P=0.846)没有显示出具有统计学意义的关系。此外,进一步行了Meta回归分析以探究异质性的来源,发现除了影响因素亚组外其余混杂因素可能不被视为异质性的来源(OS:P<0.001,DFS/RFS/PFS:P<0.001)(表2-3)。
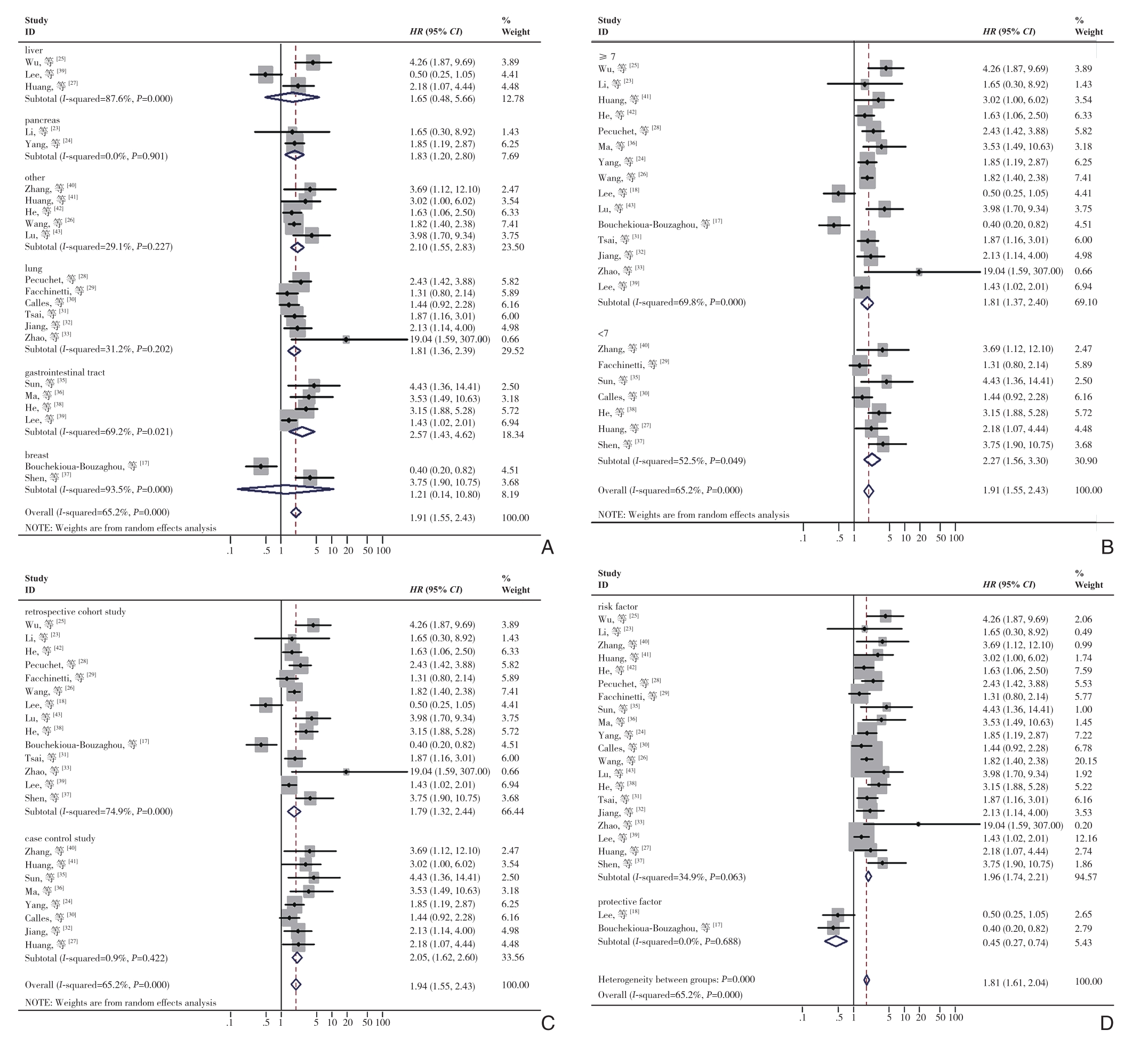
图3 针对OS混杂因素不同亚组的森林图 A:癌症类型;B:NOS评分;C:研究类型;D:影响因素
Figure 3 Forest plots of subgroups analyses of OS for different confounding factors A: Cancer types; B: NOS scores; C: Study types;D: Influential factors
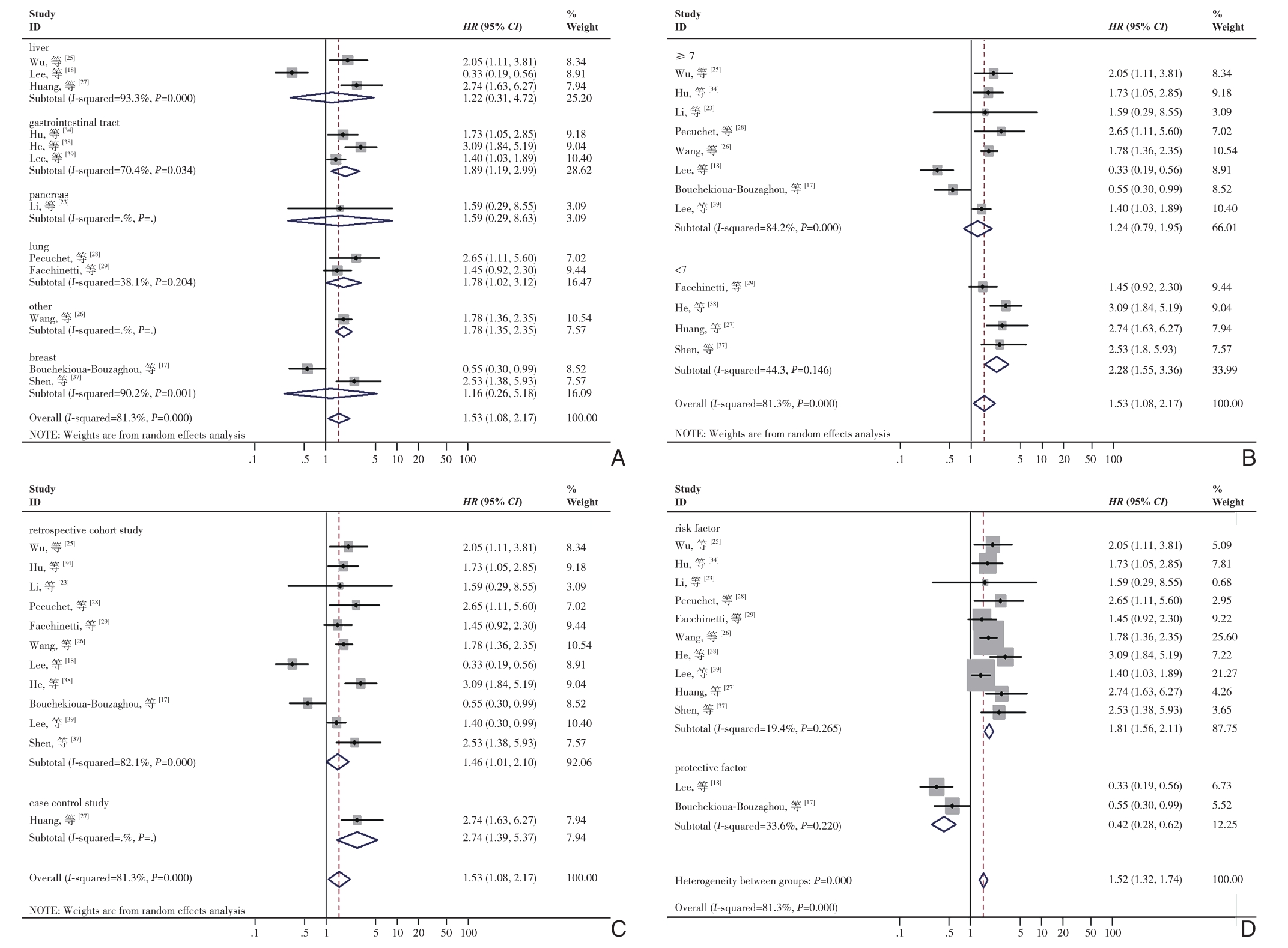
图4 针对DFS/RFS/PFS混杂因素不同亚组的森林图 A:癌症类型;B:NOS评分;C:研究类型;D:影响因素
Figure 4 Forest plots of subgroups analyses of OS for different confounding factors A: Cancer types; B: NOS scores; C: Study types;D: Influential factors
表2 LKB1表达水平的不同亚组分析的OS
Table 2 Subgroups analysis of the OS of different LKB1 expression levels
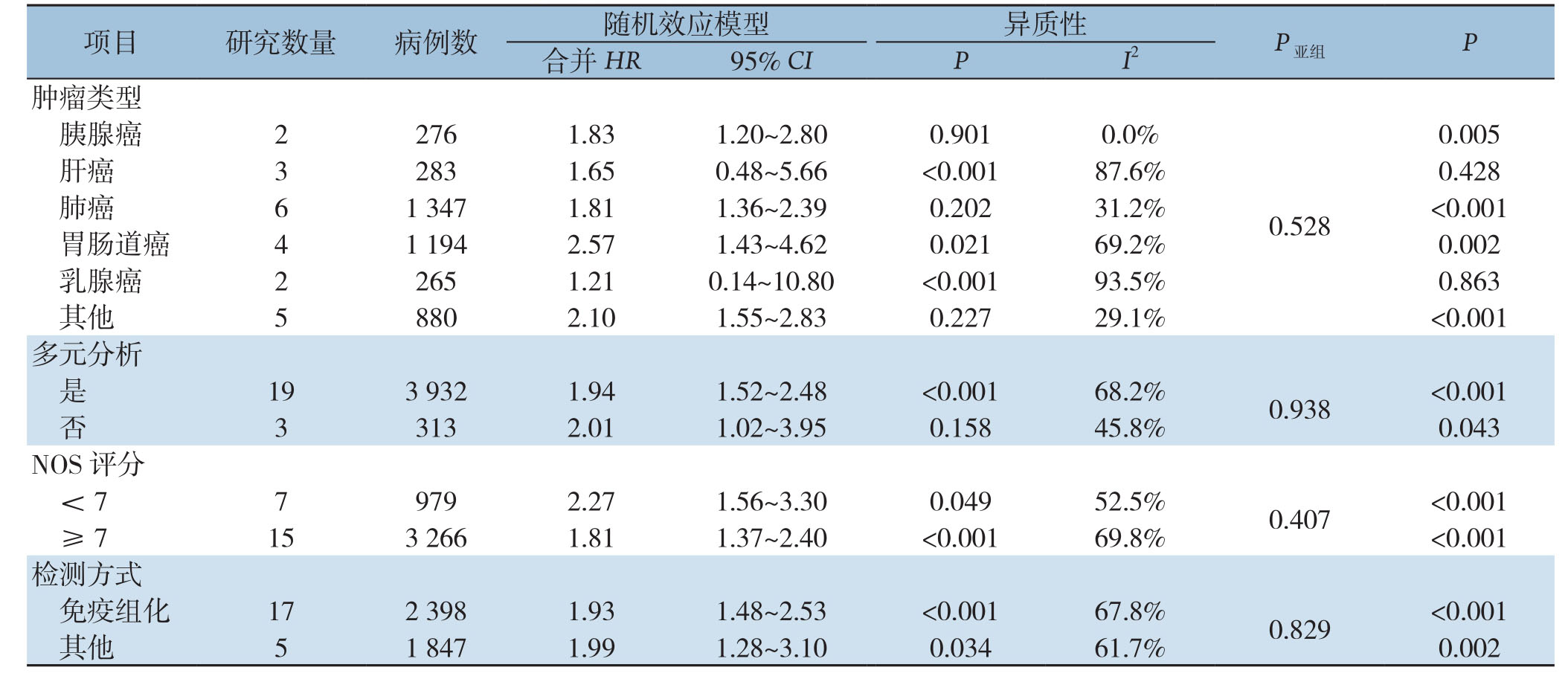
images/BZ_114_211_2285_2265_2384.png肿瘤类型胰腺癌 2 276 1.83 1.20~2.80 0.901 0.0%images/BZ_114_211_2729_2265_3173.png0.005肝癌 3 283 1.65 0.48~5.66 <0.001 87.6% 0.428肺癌 6 1 347 1.81 1.36~2.39 0.202 31.2% <0.001胃肠道癌 4 1 194 2.57 1.43~4.62 0.021 69.2% 0.002乳腺癌 2 265 1.21 0.14~10.80 <0.001 93.5% 0.863其他 5 880 2.10 1.55~2.83 0.227 29.1% <0.001 0.528
表2 LKB1表达水平的不同亚组分析的OS(续)
Table 2 Subgroups analysis of the OS of different LKB1 expression levels (continue)
images/BZ_115_211_462_2265_560.pngimages/BZ_115_211_708_2265_856.pngHR来源Cox 17 3 439 1.93 1.47~2.54 <0.001 70.1% 0.749 <0.001非 Cox 5 806 1.97 1.33~2.93 0.149 40.9% 0.001影响因素风险因素 20 3 971 2.10 1.77~2.48 0.063 34.9% <0.001 <0.001保护因子 2 274 0.45 0.27~0.74 0.688 0.0% 0.002
表3 LKB1表达水平的不同亚组分析的DFS/RFS/PFS
Table 3 Subgroups analysis of DFS/RFS/PFS of different LKB1 expression levels
images/BZ_115_211_1204_2268_1303.png癌症类型胰腺癌 1 71 1.59 0.29~8.63 — —images/BZ_115_211_1648_2268_2683.png0.591肝癌 3 283 1.22 0.31~4.72 <0.001 93.3% 0.775肺癌 2 430 1.78 1.02~3.12 0.204 38.1% 0.043胃肠道癌 3 1 037 1.89 1.19~2.99 0.034 70.4% 0.007乳腺癌 2 265 1.16 0.26~5.18 0.001 90.2% 0.846其他肿瘤 1 326 1.78 1.35~2.35 — — <0.001 0.904
2.5 LKB1表达水平在不同肿瘤类型的预后意义
为了评估LKB1表达水平在不同肿瘤类型的预后意义,进一步进行了亚组分析(图5)。对于胰腺癌,LKB1低表达与总生存率较差存在明显关系(HR=1.83,95% CI=1.20-2.80,P=0.005),而对于DFS/RFS/PFS则无统计学意义(P=0.591)。对于肝癌(OS:P=0.428;DFS/RFS/PFS,P=0.775)和乳腺癌(OS:P=0.863;DFS/RFS/PFS,P=0.846),均无统计学意义。肺癌(OS:HR=1.81,95% CI=1.36~2.39,P<0.001;DFS/RFS/PFS,HR=1.78,95% CI=1.02~3.12,P=0.043)和胃肠道癌(OS:HR=2.57,95% CI=1.43~4.62,P=0.002;DFS/RFS/PFS,HR=1.89,95% CI=1.19~2.99,P=0.007)患者中LKB1的低表达均导致预后变差。在本研究中,存在几种类型的癌症,因为其只有一项研究而被定义为其他癌症,包括皮肤恶性黑色素瘤(OS:HR=3.69,95% CI=1.12~12.10)、胶质瘤(OS:HR=3.02,95%CI=1.00~6.02)、喉癌(OS:HR=1.63,95% CI=1.06~2.50)、肝内胆管癌(OS:HR=1.82,95% CI=1.40~2.38;DFS/RFS/PFS:HR=1.78,95% CI=1.36-2.35)和前列腺癌(OS:HR=3.98,95% CI=1.70~9.34)。结果表明,其他癌症的LKB1表达水平与患者的OS和DFS/RFS/PFS也密切关系(OS:HR=2.10,95% CI=1.55~2.83,P<0.001;DFS/RFS/PFS:HR=1.78,95% CI=1.35~2.35,P<0.001)。综上所述,LKB1的表达降低引起多种肿瘤患者较差的预后。
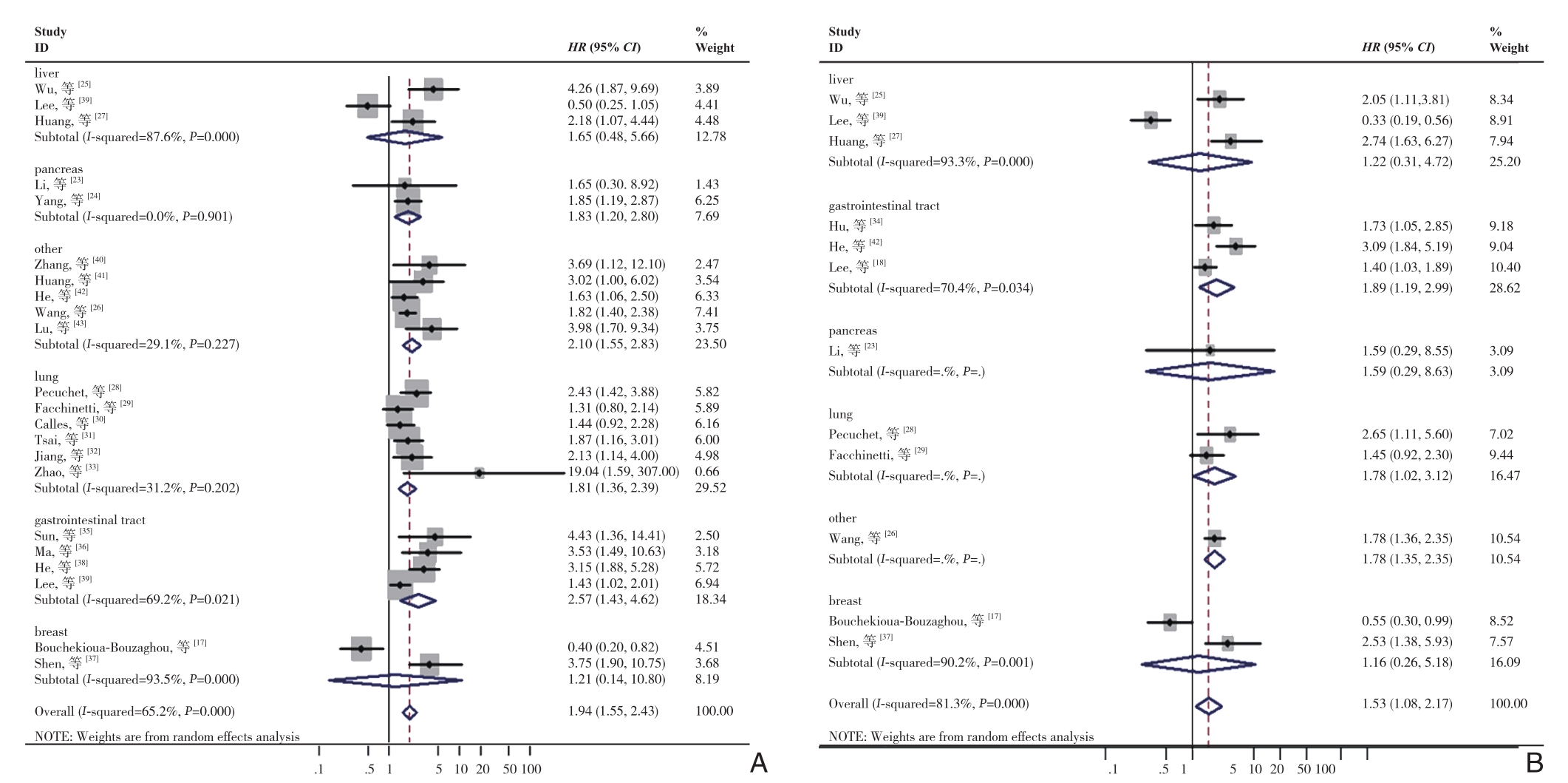
图5 LKB1表达水平与不同肿瘤类型的生存结果之间的关系分析 A:OS分析;B:DFS/RFS/PFS分析
Figure 5 Analysis of relationship between LKB1 expression levels and survival outcomes in different types cancers A: OS analysis;B: DFS/RFS/PFS analysis
2.6 发表偏倚和敏感度分析
发表偏倚是基于OS(图6A)和DFS/RFS/PFS(图6B)做出的漏斗图,结果显示没有明显的发表偏倚。敏感度分析用于评估每个研究对汇总结果的影响。所有纳入的研究都被分别排除在外,没有一项研究对OS(图7A)和DFS/RFS/PFS(图7B)的合并HR产生明显影响。
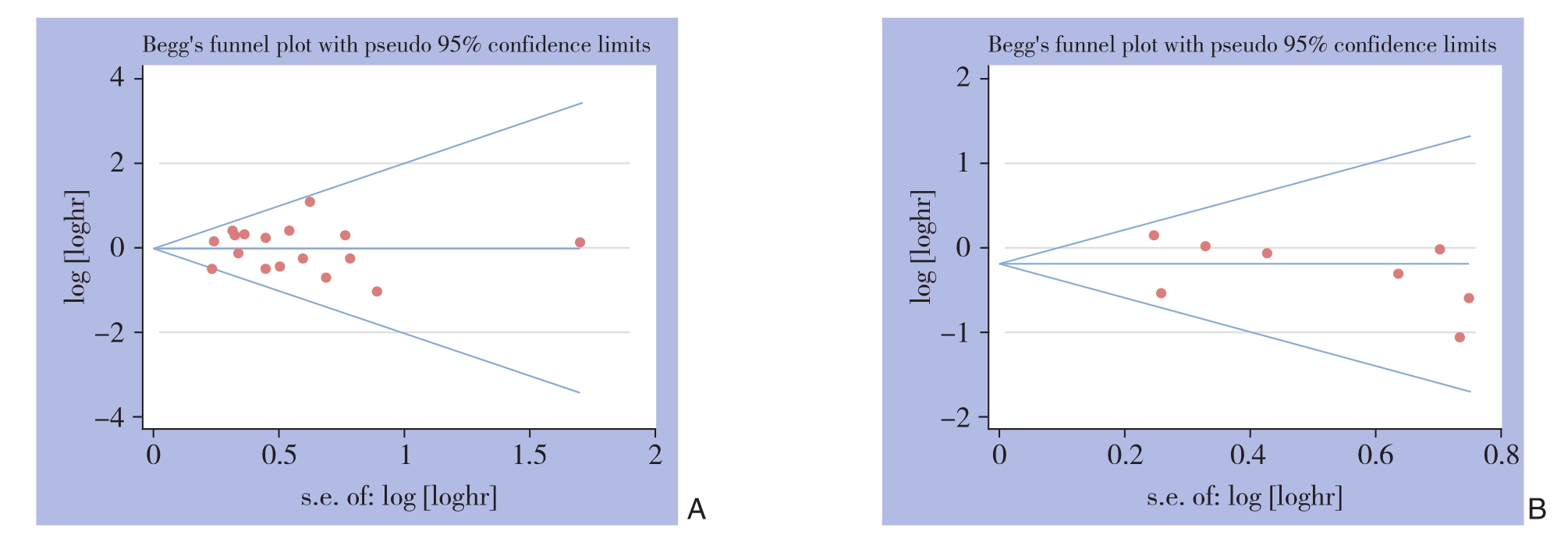
图6 Begg漏斗图 A:OS分析;B:DFS/RFS/PFS分析
Figure 6 Begg's funnel plots A: OS analysis; B: DFS/RFS/PFS analysis
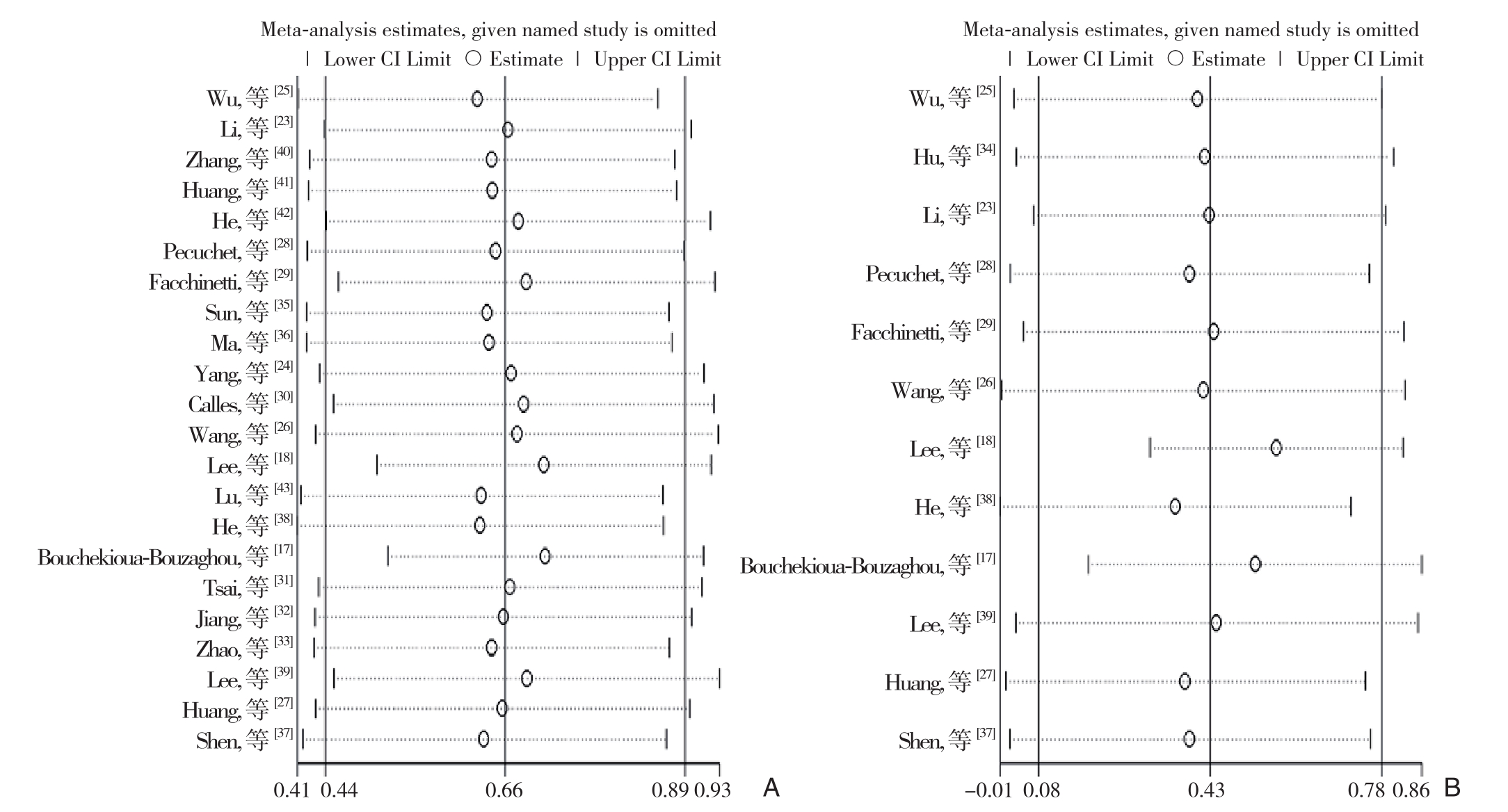
图7 所有纳入LKB1表达的预后价值的敏感性分析 A:OS分析;B:DFS/RFS/PFS分析
Figure 7 Sensitivity analysis of the prognostic value of LKB1 expression in all included studies A: OS analysis; B: DFS/RFS/PFS analysis
2.7 LKB1表达水平的生存分析
为了验证得到的结果,基因表达谱分析交互式分析(GEPIA)也用于分析患者的生存情况。通过分析高LKB1表达组的4 750例患者和低LKB1表达组的4 745例的生存情况,结果发现低LKB1表达导致了癌症患者的低生存率(OS:HR=1.14,P<0.001;DFS/RFS/PFS:HR=1.09,P=0.023)(图8)。这些数据进一步证实了本研究的准确性。
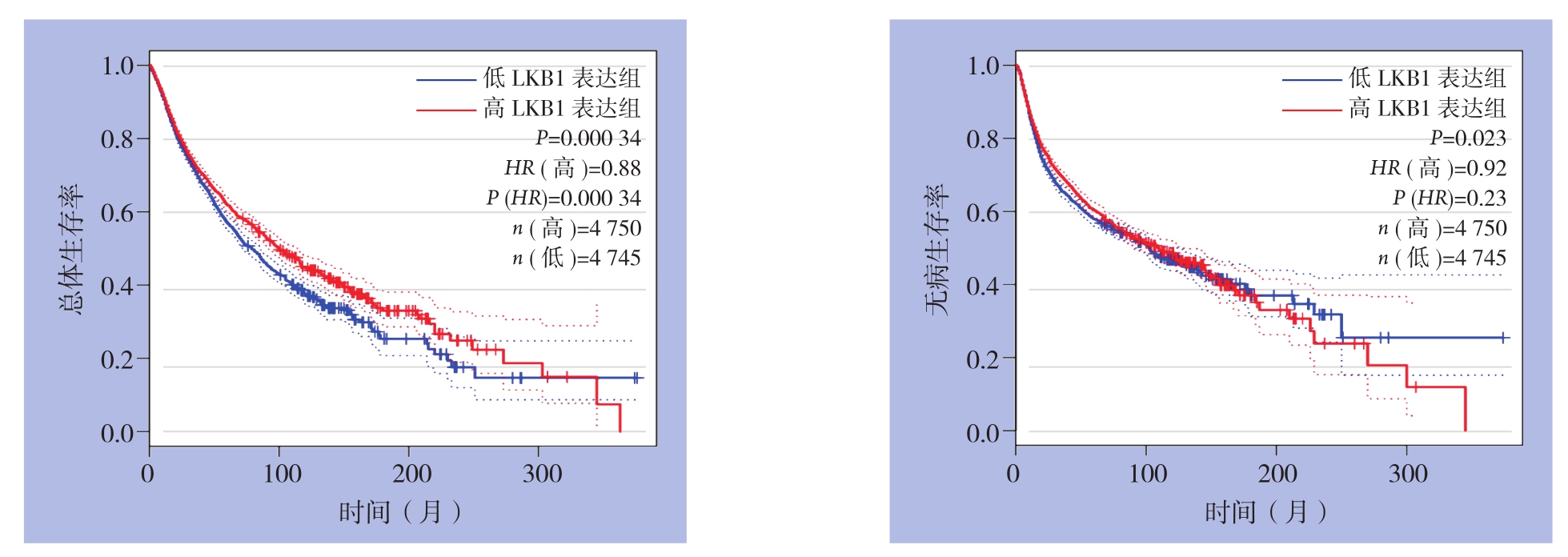
图8 GEPIA数据库显示的LKB1表达水平与生存率的关系
Figure 8 Relationship between LKB1 expression level and survival rate shown by GEPIA web tool
3 讨 论
本Meta分析汇总了先前的研究结果并增加了近几年的新研究,发现LKB1在多种肿瘤中表达降低并导致癌症患者预后变差。LKB1抑制肿瘤进展的机制极为复杂。LKB1-AMPK信号通路的激活也可以抑制癌细胞的合成代谢、调节细胞能量状态、上皮极性和细胞增殖来阻止癌细胞的生长[44-47],随着LKB1-AMPK信号通路的激活,结节性硬化复合物2(TSC2)也被该通路激活,TSC2通过抑制雷帕霉素(mTOR)及其复合物1(mTORC1)的哺乳动物靶点引起细胞自噬[48-50]。由于缺氧是大多数癌症的重要特征[51],LKB1可以调节和控制缺氧诱导因子1(HIF-1),以在缺氧条件下阻断癌细胞的增殖。此外,LKB1也是p53依赖性细胞凋亡的介体[52]并且可以通过激活JNK途径诱导细胞凋亡[53]。
尽管已经对LKB1进行了Meta分析和系统评估,但笔者在已发表的Meta分析中发现了一些问题。首先,先前的Meta分析中纳入的文献只有14篇,并且在2015年之前发表。而且通过使用更全面的检索策略,发现了2015年之前发表的3篇文章具有误导性[33,39,43],这些文章报道了KRAS突变联合LKB1低表达以及LKB1突变与肿瘤患者不良预后的相关性,明显纳入了混杂因素。并且2016年以来发布的几份新的研究可能会对以往的结论产生一定的影响,因此有必要重新分析LKB1表达与生存之间的相关性。其次,由于使用DFS/RFS/PFS作为结局变量的论文数量太少,无法在已发表的Meta分析中得出LKB1与该预后指标之间的结论,而我们的结果有望重新发现这一点。第三,以往的Meta分析仅包括回顾性队列研究。而病例对照研究也包括在本Meta分析中,并用于探索异质性的来源。第四,异质性的来源存在问题,应该根据相关变量将研究划分为亚组之后分析。
本研究结果表明,LKB1表达的下降与肿瘤患者的OS和DFS/RFS/PFS较差明显有关。然后通过进行亚组分析,并根据癌症类型、NOS得分、研究类型和影响因素来讨论异质性的来源,结果发现,将所有纳入的文章根据风险或保护因素分为两个亚组时,两部分的潜在异质性明显降低,这意味着风险或保护性因素可以部分解释OS和DFS/RFS/PFS中的异质性来源。虽然其他伴随变量并未显示出异质性的降低,在执行Meta回归后,无论是OS还是DFS/RFS/PFS,只有在影响因素P<0.05的亚组中,才能进一步验证异质性来源的分析。本研究结果还表明,LKB1与预后结果之间的相关性在不同类型的肿瘤中发生了变化。在本研究无明显的出版偏倚,结果可以被认为是稳定的。敏感性分析结果显示在所有纳入的研究被逐一地排除之后,没有一个对OS和DFS/RFS/PFS的合并HR产生明显影响。考虑到这些方面,LKB1低表达可以导致较差的生存结局,LKB1表达水平可以被视为有价值的肿瘤预后独立预测因子和治疗靶标。
与先前的Meta分析一样,有2篇文章[17-18]显示了相反的结果。在Lee等[18]的研究中指出,癌细胞的存活需要激活Ras/Skp2/LKB1信号通路,即Skp2介导的LKB1多聚泛素化和复合物形成。而在Bouchekioua-Bouzaghou的研究中指出,细胞质中高水平的LKB1导致乳腺肿瘤细胞预后不良,亚细胞定位可能是产生相反结果的主要原因[17]。除此之外,研究者[54]还发现LKB1在肝癌细胞中下调可能引起其对5-氟尿嘧啶的耐药,所以更需要进一步研究去说明LKB1在肿瘤中的复杂作用。
不过,本研究同样存在局限性。首先,纳入文献中统计异质性相对较大。异质性的来源可能多种多样,包括患者的临床特征、LKB1的检测技术、样品的类型、纳入研究的方法等,而风险或保护因素只是异质性来源的一部分。第二,与癌症进展有关的因素很多,但由于篇幅有限,因此没有进行性别、年龄和其他类型的亚组分析。第三,在大多数文献中,利用免疫组织化学(IHC)检测LKB1蛋白的表达量,但尚无确定降低IHC的适当临界值以及LKB1表达降低的标准。第四,没有收集证据来证明LKB1表达与血液系统恶性肿瘤之间的关系。第五,几篇文章仅提供了Kaplan-Meier曲线,通过追踪点计算的HR和95% CI可能或多或少地存在偏差。最后,由于本文中评估的是多种肿瘤类型中低表达的LKB1与患者不良预后之间的关系,这也可能会导致结论的不准确性及摇摆性。
本研究证实了低表达的LKB1与多种类型肿瘤患者预后较差明显相关,LKB1有可能作为有价值的独立预测指标和治疗靶标,具有较高的临床应用价值。同时,本研究的结论需要更多后续的研究去证实。
[1] Bray F, Ferlay J, Soerjomataram I, et al.Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J].CA Cancer J Clin, 2018,68(6):394-424.doi: 10.3322/caac.21492.
[2] Mehenni H, Gehrig C, Nezu J, et al.Loss of LKB1 kinase activity in Peutz-Jeghers syndrome, and evidence for allelic and locus heterogeneity[J].Am J Hum Genet, 1998, 63(6):1641-1650.doi:10.1086/302159.
[3] Hemminki A, Tomlinson I, Markie D, et al.Localization of a susceptibility locus for Peutz-Jeghers syndrome to 19p using comparative genomic hybridization and targeted linkage analysis[J].Nat Genet, 1997, 15(1):87-90.doi: 10.1038/ng0197-87.
[4] Alessi DR, Sakamoto K, Bayascas JR.LKB1-dependent signaling pathways[J].Annu Rev Biochem, 2006,75:137-163.doi: 10.1146/annurev.biochem.75.103004.142702.
[5] Liu Y, Marks K, Cowley GS, et al.Metabolic and functional genomic studies identify deoxythymidylate kinase as a target in LKB1-mutant lung cancer[J].Cancer Discov, 2013, 3(8):870-879.doi: 10.1158/2159-8290.CD-13-0015.
[6] Ji H, Ramsey MR, Hayes DN, et al.LKB1 modulates lung cancer differentiation and metastasis[J].Nature, 2007, 448(7155):807-810.doi: 10.1038/nature06030.
[7] Faubert B, Vincent EE, Griss T, et al.Loss of the tumor suppressor LKB1 promotes metabolic reprogramming of cancer cells via HIF-1α[J].Proc Natl Acad Sci U S A, 2014, 111(7):2554-2559.doi:10.1073/pnas.1312570111.
[8] Gao Y, Xiao Q, Ma H, et al.LKB1 inhibits lung cancer progression through lysyl oxidase and extracellular matrix remodeling[J].Proc Natl Acad Sci U S A, 2010, 107(44):18892-18897.doi: 10.1073/pnas.1004952107.
[9] Chan KT, Asokan SB, King SJ, et al.LKB1 loss in melanoma disrupts directional migration toward extracellular matrix cues[J].J Cell Biol, 2014, 207(2):299-315.doi: 10.1083/jcb.201404067.
[10] Gan RY, Li HB.Recent progress on liver kinase B1 (LKB1):expression, regulation, downstream signaling and cancer suppressive function[J].Int J Mol Sci, 2014, 15(9):16698-16718.doi: 10.3390/ijms150916698.
[11] Etienne-Manneville S.Polarity proteins in migration and invasion[J].Oncogene, 2008, 27(55):6970-6980.doi:10.1038/onc.2008.347.
[12] Martin-Belmonte F, Perez-Moreno M.Epithelial cell polarity,stem cells and cancer[J].Nat Rev Cancer, 2011, 12(1):23-38.doi:10.1038/nrc3169.
[13] Sanchez-Cespedes M.A role for LKB1 gene in human cancer beyond the Peutz-Jeghers syndrome[J].Oncogene, 2007,26(57):7825-7832.doi: 10.1038/sj.onc.1210594.
[14] Murray CW, Brady JJ, Tsai MK, et al.An LKB1-SIK Axis Suppresses Lung Tumor Growth and Controls Differentiation[J].Cancer Discov, 2019, 9(11):1590-1605.doi: 10.1158/2159-8290.CD-18-1237.
[15] Kuwako KI, Okano H.The LKB1-SIK Pathway Controls Dendrite Self-Avoidance in Purkinje Cells[J].Cell Rep, 2018, 24(11):2808-2818.doi: 10.1016/j.celrep.2018.08.029.
[16] Wang Y, Du X, Wei J, et al.LKB1 orchestrates dendritic cell metabolic quiescence and anti-tumor immunity[J].Cell Res, 2019,29(5):391-405.doi: 10.1038/s41422-019-0157-4.
[17] Bouchekioua-Bouzaghou K, Poulard C, Rambaud J, et al.LKB1 when associated with methylatedERα is a marker of bad prognosis in breast cancer[J].Int J Cancer, 2014, 135(6):1307-1318.doi:10.1002/ijc.28781.
[18] Lee SW, Li CF, Jin G, et al.Skp2-dependent ubiquitination and activation of LKB1 is essential for cancer cell survival under energy stress[J].Mol Cell, 2015, 57(6):1022-1033.doi: 10.1016/j.molcel.2015.01.015.
[19] Xiao J, Zou Y, Chen X, et al.The Prognostic Value of Decreased LKB1 in Solid Tumors: A Meta-Analysis[J].PLoS One, 2016,11(4):e0152674.doi: 10.1371/journal.pone.0152674.
[20] Liberati A, Altman DG, Tetzlaff J, et al.The PRISMA statement for reporting systematic reviews and meta-analyses of studies that evaluate health care interventions: explanation and elaboration[J]Ann Intern Med, 2009, 151(4):W65-94.doi: 10.7326/0003-4819-151-4-200908180-00136.
[21] Stang A.Critical evaluation of the Newcastle-Ottawa scale for the assessment of the quality of nonrandomized studies in metaanalyses[J].Eur J Epidemiol, 2010, 25(9):603-605.doi: 10.1007/s10654-010-9491-z.
[22] Greenland S.Quantitative methods in the review of epidemiologic literature[J].Epidemiol Rev, 1987, 9:1-30.
[23] Li D, Zhou Y, Liu Y, et al.Decreased expression of LKB1 predicts poor prognosis in pancreatic neuroendocrine tumor patients undergoing curative resection[J].Onco Targets Ther, 2018,11:1259-1265.doi: 10.2147/OTT.S154168.
[24] Yang JY, Jiang SH, Liu DJ, et al.Decreased LKB1 predicts poor prognosis in Pancreatic Ductal Adenocarcinoma[J].Sci Rep, 2015,5:10575.doi: 10.1038/srep10575.
[25] Wu CC, Wu DW, Lin YY, et al.Hepatitis B virus X protein represses LKB1 expression to promote tumor progression and poor postoperative outcome in hepatocellular carcinoma[J].Surgery,2018, 163(5):1040-1046.doi: 10.1016/j.surg.2017.11.030.
[26] Wang J, Zhang K, Wang J, et al.Underexpression of LKB1 tumor suppressor is associated with enhanced Wnt signaling and malignant characteristics of human intrahepatic cholangiocarcinoma[J].Oncotarget, 2015, 6(22):18905-18920.doi: 10.18632/oncotarget.4305.
[27] Huang YH, Chen ZK, Huang KT, et al.Decreased expression of LKB1 correlates with poor prognosis in hepatocellular carcinoma patients undergoing hepatectomy[J].Asian Pac J Cancer Prev, 2013,14(3):1985-1988.doi: 10.7314/apjcp.2013.14.3.1985.
[28] Pécuchet N, Laurent-Puig P, Mansuet-Lupo A, et al.Different prognostic impact of STK11 mutations in non-squamous non-smallcell lung cancer[J].Oncotarget, 2017, 8(14):23831-23840.doi:10.18632/oncotarget.6379.
[29] Facchinetti F, Bluthgen MV, Tergemina-Clain G, et al.LKB1/STK11 mutations in non-small cell lung cancer patients: Descriptive analysis and prognostic value[J].Lung Cancer, 2017, 112:62-68.doi: 10.1016/j.lungcan.2017.08.002.
[30] Calles A, Sholl LM, Rodig SJ, et al.Immunohistochemical Loss of LKB1 Is a Biomarker for More Aggressive Biology in KRAS-Mutant Lung Adenocarcinoma[J].Clin Cancer Res, 2015,21(12):2851-2860.doi: 10.1158/1078-0432.CCR-14-3112.
[31] Tsai LH, Chen PM, Cheng YW, et al.LKB1 loss by alteration of the NKX2-1/p53 pathway promotes tumor malignancy and predicts poor survival and relapse in lung adenocarcinomas[J].Oncogene,2014, 33(29):3851-3860.doi: 10.1038/onc.2013.353.
[32] Jiang L, Liang X, Liu M, et al.Reduced expression of liver kinase B1 and Beclin1 is associated with the poor survival of patients with non-small cell lung cancer[J].Oncol Rep, 2014, 32(5):1931-1938.doi: 10.3892/or.2014.3432.
[33] Zhao N, Wilkerson MD, Shah U, et al.Alterations of LKB1 and KRAS and risk of brain metastasis: comprehensive characterization by mutation analysis, copy number, and gene expression in nonsmall-cell lung carcinoma[J].Lung Cancer, 2014, 86(2):255-261.doi: 10.1016/j.lungcan.2014.08.013.
[34] Hu M, Zhao T, Liu J, et al.Decreased expression of LKB1 is associated with epithelial-mesenchymal transition and led to an unfavorable prognosis in gastric cancer[J].Hum Pathol, 2019,83:133-139.doi: 10.1016/j.humpath.2018.08.017.
[35] Sun J, Ling B, Xu X, et al.Decreased Expression of Tumorsuppressor Gene LKB1 Correlates with Poor Prognosis in Human Gastric Cancer[J].Anticancer Res, 2016, 36(3):869-875.
[36] Ma LG, Bian SB, Cui JX, et al.LKB1 inhibits the proliferation of gastric cancer cells by suppressing the nuclear translocation of Yap and β-catenin[J].Int J Mol Med, 2016, 37(4):1039-1048.doi:10.3892/ijmm.2016.2494.
[37] Shen Z, Wen XF, Lan F, et al.The tumor suppressor gene LKB1 is associated with prognosis in human breast carcinoma[J].Clin Cancer Res, 2002, 8(7):2085-2090.
[38] He TY, Tsai LH, Huang CC, et al.LKB1 loss at transcriptional level promotes tumor malignancy and poor patient outcomes in colorectal cancer[J].Ann Surg Oncol, 2014, 21(Suppl 4):S703-710.doi:10.1245/s10434-014-3824-1.
[39] [39] Lee SJ, Kang BW, Chae YS, et al.Genetic variations in STK11,PRKAA1, and TSC1 associated with prognosis for patients with colorectal cancer[J].Ann Surg Oncol, 2014, 21(Suppl 4):S634-639.doi: 10.1245/s10434-014-3729-z.
[40] Zhang W, Li X, Song G, et al.Prognostic significance of LKB1 promoter methylation in cutaneous malignant melanoma[J].Oncol Lett, 2017, 14(2):2075-2080.doi: 10.3892/ol.2017.6431.
[41] Huang J, Chen H, Wei Q, et al.Downregulation of LKB1 promotes tumor progression and predicts unfavorable prognosis in patients with glioma[J].Oncol Lett, 2017, 13(3):1688-1694.doi: 10.3892/ol.2017.5631.
[42] He SS, Chen Y, Wang HZ, et al.Loss of LKB1 Expression Decreases the Survival and Promotes Laryngeal Cancer Metastasis[J].J Cancer, 2017, 8(17):3548-3554.doi: 10.7150/jca.20535.
[43] Lu J, Sun P, Sun B, et al.Low LKB1 Expression Results in Unfavorable Prognosis in Prostate Cancer Patients[J].Med Sci Monit, 2015, 21:3722-3727.doi: 10.12659/msm.894847.
[44] Hardie DG, Alessi DR.LKB1 and AMPK and the cancermetabolism link - ten years after[J].BMC Biol, 2013, 11:36.doi:10.1186/1741-7007-11-36.
[45] Lee JH, Koh H, Kim M, et al.Energy-dependent regulation of cell structure by AMP-activated protein kinase[J].Nature, 2007,447(7147):1017-1020.doi: 10.1038/nature05828.
[46] Forcet C, Billaud M.Dialogue between LKB1 and AMPK: a hot topic at the cellular pole[J].Sci STKE, 2007, 2007(404):pe51.doi:10.1126/stke.4042007pe51.
[47] Ciccarese F, Zulato E, Indraccolo S.LKB1/AMPK Pathway and Drug Response in Cancer: A Therapeutic Perspective[J].Oxid Med Cell Longev, 2019, 2019:8730816.doi: 10.1155/2019/8730816.
[48] Shaw RJ.LKB1 and AMP-activated protein kinase control of mTOR signaling and growth[J].Acta Physiol (Oxf), 2009,196(1):65-80.doi: 10.1111/j.1748-1716.2009.01972.x.
[49] Jia J, Abudu YP, Claude-Taupin A, et al.Galectins control MTOR and AMPK in response to lysosomal damage to induce autophagy[J].Autophagy, 2019, 15(1):169-171.doi:10.1080/15548627.2018.1505155.
[50] Han Y, Feng H, Sun J, et al.Lkb1 deletion in periosteal mesenchymal progenitors induces osteogenic tumors through mTORC1 activation[J].J Clin Invest, 2019, 129(5):1895-1909.doi:10.1172/JCI124590.
[51] Wilson WR and Hay MP.Targeting hypoxia in cancer therapy[J].Nat Rev Cancer, 2011, 11(6):393-410.doi: 10.1038/nrc3064.
[52] Karuman P, Gozani O, Odze RD, et al.The Peutz-Jegher gene product LKB1 is a mediator of p53-dependent cell death[J].Mol Cell, 2001, 7(6):1307-1319.doi: 10.1016/s1097-2765(01)00258-1.
[53] Lee JH, Koh H, Kim M, et al.JNK pathway mediates apoptotic cell death induced by tumor suppressor LKB1 in Drosophila[J].Cell Death Differ, 2006, 13(7):1110-1122.doi: 10.1038/sj.cdd.4401790.
[54] 叶劲松.RSK2沉默对肝癌细胞化疗药物敏感性的影响[J].中国普通外科杂志, 2012, 21(12):1546-1549.Ye JS.RSK2 silencing enhances the sensitivity of hepatocellular carcinoma cells to chemotherapeutic agents[J].Chinese Journal of General Surgery, 2012, 21(12):1546-1549.
