胃癌(gastric cancer,GC)是人类消化道中最常见的恶性肿瘤之一[1],根据全球癌症统计分析数据,GC已成为第五大最常被诊断出的癌症和 第三大癌症致死的原因,是全球性的重要健康危 机[2]。临床上以手术切除、化疗、放疗或联合靶向为主的GC治疗方法难以彻底清除肿瘤病灶,肿瘤易进展或复发且毒副作用大,患者的5年生存率低至10%~15%[3-4],因此探索新的更有效的治疗方法成为亟待解决的重大课题。
近年来肿瘤免疫治疗作为一种新型的治疗方法,基于人体的免疫系统,利用免疫调节发挥抗肿瘤作用,已经表现出显著的临床效果瘤生长、发展和患者预后结局均与肿瘤免疫细胞浸润情况相关[5-7]。2018年,James P.Allison和Tasuku Honjo 等就因在“抑制免疫检查点(CTLA-4、PD-1)抗肿瘤研究”中作出卓越贡献而获得诺贝尔生理学奖。此外,已有研究表明,M2巨噬细胞在膀胱癌组织中富集,促进血管生成,可作为膀胱癌潜在的免疫治疗靶点[8],有关自然杀伤细胞的多种肿瘤免疫治疗也已进入临床试验阶段[9]。由此可见,挖掘免疫细胞相关靶标是优化肿瘤免疫治疗的有效路径[10-11]。
过去这方面的研究多通过流式细胞术或免疫组织化学来评估肿瘤中浸润的免疫细胞的组成,但这些方法都存在不足,并有可能导致细胞丢失或结果失真。Newman等[12]于2015年设计出了新的生物信息学算法—CIBERSORT,通过估计RNA转录本的相对子集和复杂组织标准化基因表达数据来鉴定细胞类型和被测样本的免疫细胞组成。该法已经在多种恶性肿瘤中进行了很好的验证[13],可以广泛地应用于恶性肿瘤发病机制、肿瘤免疫等过程的生物信息学挖掘。另有研究表明,免疫治疗与化疗联合在多种肿瘤治疗中效果显著,寻找肿瘤免疫的基因标志物已经成为当下肿瘤治疗的研究热点[14-15]。而作为鉴定与疾病相关的基因模块和关键基因的理想方法,加权基因共表达网络分析技术(weighted gene co-expression network analysis,WGCNA)基于大样本基因表达数据寻找潜在生物标志物,已经在众多研究中被用来对宫颈癌、甲状腺乳头状癌等恶性肿瘤进行分析,并成功筛选出了有效的基因靶点[16-17]。
综上,为寻找可靠的GC相关免疫细胞相关基因靶标,从而优化GC患者的预后,本研究利用TCGA数据库下载GC的mRNA表达数据,并使用CIBERSORT算法逐一测算样品的免疫细胞组成,构建WGCNA,结合生存分析获取其与GC预后的相关性并用外部数据库加以验证,鉴定出GC免疫细胞浸润相关预后基因,为进一步明确潜在的GC免疫治疗靶点提供理论依据。
1 资料与方法
1.1 数据的采集
本研究从TCGA数据库中下载了共407例基因转录表达谱数据,其中包括癌组织样本375例,癌旁组织样本32例。GC样本临床信息包含性别、生存时间、生存状态、临床分期、肿瘤分级等。人类癌症标本核心资源库的科研人员在采集、处理和分派癌组织标本和癌旁组织标本时,去除了患者的身份信息,因此不存在伦理问题。
1.2 肿瘤浸润免疫细胞评估
利用R语言的limma包,对GC组织及正常组织转录本的mRNA表达谱数据进行校正。肿瘤浸润免疫细胞的评估采用CIBERSORT算法,反卷积法处理标记基因表达值,估算肿瘤组织中免疫细胞占比。这些免疫细胞包括M0 巨噬细胞、浆细胞、静止记忆性CD4 T细胞、CD8 T细胞、活化记忆性CD4 T细胞、调节型T细胞、记忆性B细胞等 22种。为提高准确度,以P<0.05为标准对样本进行筛选,绘制所有符合条件的样本中每种免疫细胞占比柱状图,免疫细胞间的相关性热图和GC组织与正常组织样本免疫细胞占比的小提琴图。
1.3 肿瘤组织中高含量免疫细胞的生存分析
结合从TCGA下载的患者临床信息,合并免疫细胞矩阵和生存时间,对在肿瘤组织中含量显著偏高的11种免疫细胞进行批量化生存分析。根据免疫细胞水平中位值将患者分为高、低浸润组,采用Kaplan-Meier法计算其与生存的相关性,使用R语言的survival包绘制生存曲线。
1.4 WGCNA 模块构建及可视化
WGCNA技术是一种为分析基因表达数据而建立的高通量数据挖掘算法。相较于一维分子生物学的研究方法,其构建的基因模块几乎覆盖了人类的所有基因,对生物系统的展示更加精准。本研究利用R语言中WGCNA包进行了基因共表达网络的构建与模块鉴定。首先将数据导入R语言进行初步筛选,去除离群值以保证可信度,然后绘制样本层次聚类树,并依据差异基因表达谱定义相似度表达矩阵,对基因进行相似性度量。
具体而言,用Sij表示基因i和基因j的表达相似度,通过皮尔森相关系数(Pearson correlation coefficient,Pcc)计算获得相关系数S i j=|cor(i,j)|。由此构成相似度矩阵S=[Sij]。对任意的两个基因i、j之间的关联,它们的临接系数aij为aij= power(Xij ,β)≡|Xij|β,即对相关系数进行次方的幂指数加权,得到软阈值β。对最佳β值的选择,应当使得R2值趋向于稳定且R2>0.8[18]。既遵循无尺度网络原则,又确保不同模块中基因间链接程度较高,以包含足够的生物信息。采用拓扑重叠技术(Topological Overlap Measure, TOM)对衡量基因间的关联性,将邻接矩阵|Xij|转换为拓扑矩阵Ω=|ωij|[19]。基因i和基因j之间若无直接相关关系,且又无第三方基因将这两个基因间接连接起来,即视为两个基因之间相关性为0(TOM=0),两基因之间的差异度为(1-TOM)。基因之间的相关性不仅包括两个相关系数,还包括第三方基因建立的相关性,所得结果与实际情况更加吻合,为共表达网络的构建奠定了基础。
基于蛋白质互作网络的算法来完成共表达网络中模块的识别。将每个模块中基因最小数目定义为5 0,应用基于层次的模块识别,以差异度值(1-TOM)为基础,使用动态剪接法和聚类分析探测基因的功能模块。基因由树中的叶节点代表,密集连接的分支代表了接近的基因,函数“moduleEigengenes”计算得到的模块特征基因(module eigengene,ME)为模块的第一主成分,即该模块基因表达谱代表。若模块间的ME相关性>0.75,则表示它们有类似的表达谱,应将该模块合并。将合并后的模块与免疫细胞建立关联分析,绘制模块—免疫细胞相关热图,根据模块中每个基因的基因与模块之间相关性值(module membership,MM)和基因与免疫细胞之间的相关性绝对值(gene significance,GS)得到与预后免疫细胞相关性最显著的核心模块。
1.5 核心模块中差异基因的生存分析
利用R语言survival包,分别对与静止记忆性CD4 T细胞和调节型T细胞相关的核心模块中的差异基因进行生存分析。依据GC组织中差异基因表达量中位数作为分界,根据基因表达量将患者分为高低表达组,采用Kaplan-Meier、Log-rank法计算基因与患者总生存期的相关性,并使用R语言的survival包进行生存曲线绘制,根据计算出的P<0.05筛选差异生存基因。
1.6 基于外部数据库验证差异生存基因的预后意义
Kaplan-Meier plotter数据库是进行生存分析最权威的网站之一,包含了多个平台的GC 数据,基于在线Kaplan-Meier plotter数据库,验证数据库中包含的关键基因的预后效能。通过NCBI(https://www.ncbi.nlm.nih.gov/)下载了GSE84437,GSE57303,GSE62254等多组GEO数据库中独立数据集来验证关键基因的预后 意义。
1.7 差异生存基因的通路富集分析
为了注释和分析生存差异基因的生物学功能,应用基因本体论富集分析(geneontology enrichment analysis,GO)、京都基因和基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)进行途径注释和可视化,对P<0.05的富集结果予以保留。其中GO分析分别选取生物过程(biological process,BP),细胞成分(cell component,CC)和分子功能(molecular function,MF)3大类展示富集结果;KEGG利用其全面的数据库资源,对基因信息对更高层次和更复杂细胞活动以及生物体行为做出量化推测。
2 结 果
2.1 GC 中浸润免疫细胞情况
在获得的所有样本中,有258例GC样本和18例正常样本符合CIBERSORT标准,即P<0.05。在应用CIBERSORT算法分析数据后,利用R语言作图。首先,用一种颜色代表一种免疫细胞类型,用柱形的长度代表一种类型免疫细胞的相对含量,各样本不同类型免疫细胞的相对含量之和为1,绘制出了22种免疫细胞在每个样本中的柱状占比图。结果发现,巨噬细胞(M0)、中性粒细胞、活化的CD4+记忆性T细胞、静止记忆性CD4 T细胞、调节型T细胞等11种免疫细胞在肿瘤组织中明显高于正常组织(图1)。使用Pearson相关分析计算258例GC样本中22种免疫细胞浸润的相关度,绘制出每对免疫细胞间的相关性热图,红色代表协同,蓝色代表拮抗,相关性随着颜色的加深而变高。在GC组织中,构成比相关系数较大的免疫细胞包括:呈高负相关性的静止记忆性CD4 T细胞与CD8 T细胞(r=-0.51),呈高正相关性的活化记忆性CD4 T细胞与CD8 T细胞(r=0.42)等(图2)。用红色和蓝色代表GC组织和正常组织,横坐标表示免疫细胞,纵坐标表示免疫细胞浸润的百分比,绘制GC及正常组织中免疫细胞浸润占比小提琴图。结果发现,258例GC样本和18例正常样本的浆细胞、静息状态记忆性CD4 T细胞、活化状态记忆性CD4 T细胞及辅助性T细胞等浸润程度差异均具有统计学意义(P<0.001、P=0.013、P=0.001、P=0.012),其中浆细胞在正常样本组中的浸润程度较GC样本组高,而静息状态记忆性CD4 T细胞、活化记忆性CD4 T细胞及辅助性T细胞正常样本组中的浸润程度低于GC样本组(图3)。
2.2 GC 免疫细胞浸润与预后的关系
对于上述在肿瘤组织中含量显著高于正常组织的11种免疫细胞,根据免疫细胞浸润值将患者分为高、低浸润组。采用Kaplan-Meier与Log-rank法进行生存分析。最后发现静止记忆性CD4 T细胞和调节型T细胞的浸润情况与GC患者生存率呈明显相关(P=0.021、P=0.013)。其中静止记忆性CD4 T细胞构成比例高者生存期短,而调节型T细胞构成比例高者生存期长(图4)。
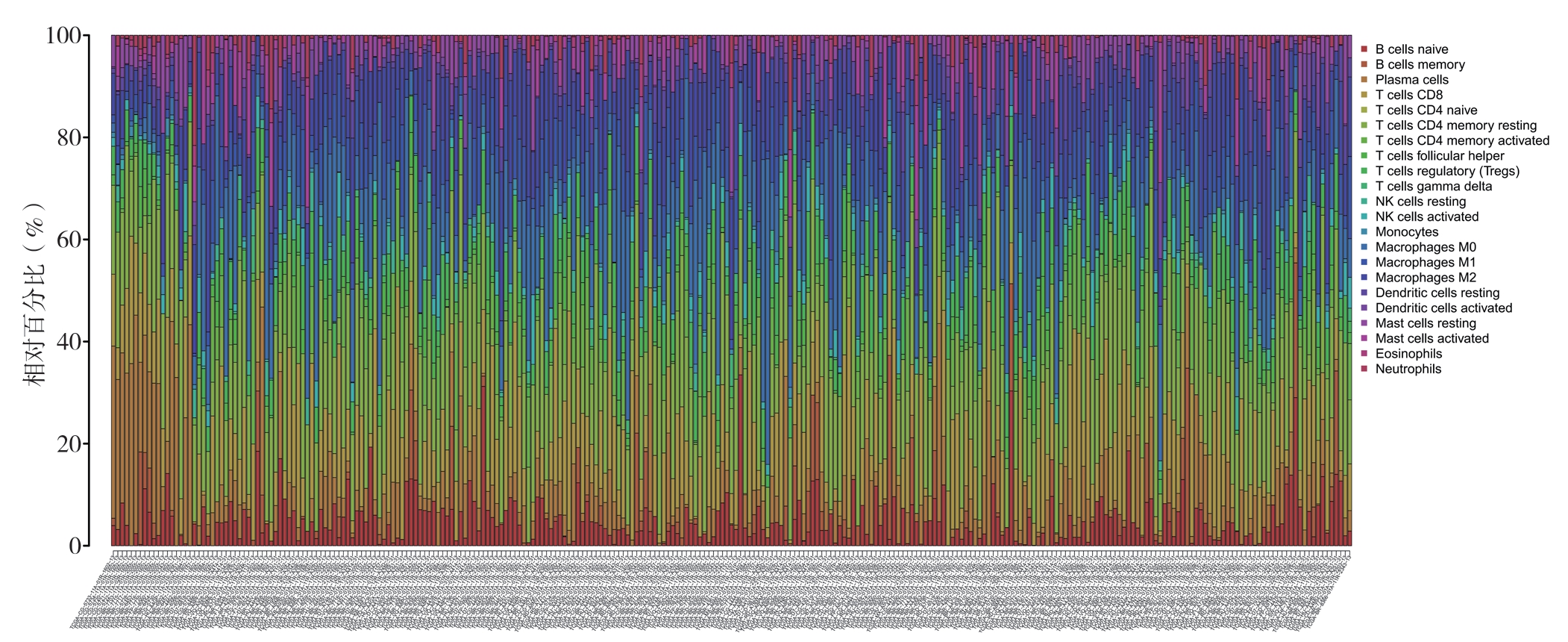
图1 所有标本中22 种免疫细胞占比的柱状图
Figure 1 Histogram of the proportions of 22 immune cells in all specimens
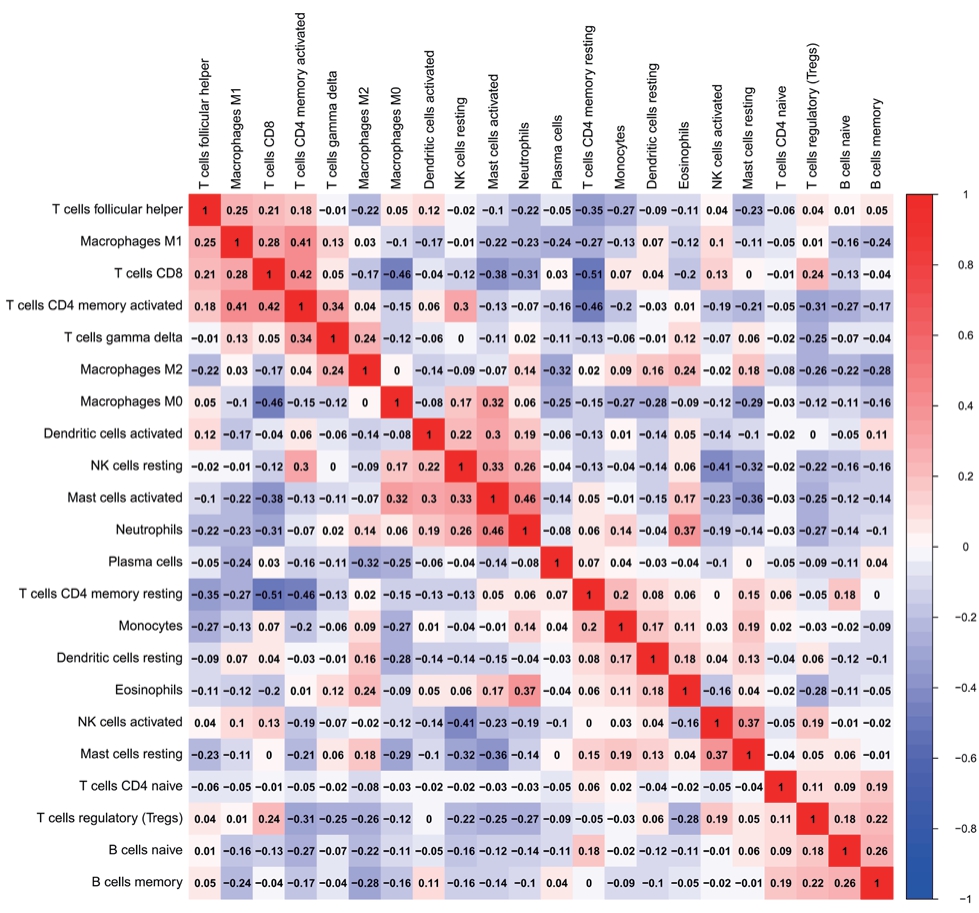
图2 GC 组织样本中每种免疫细胞占比相关图
Figure 2 Correlation diagram of the proportion of each immune cell in GC tissue samples
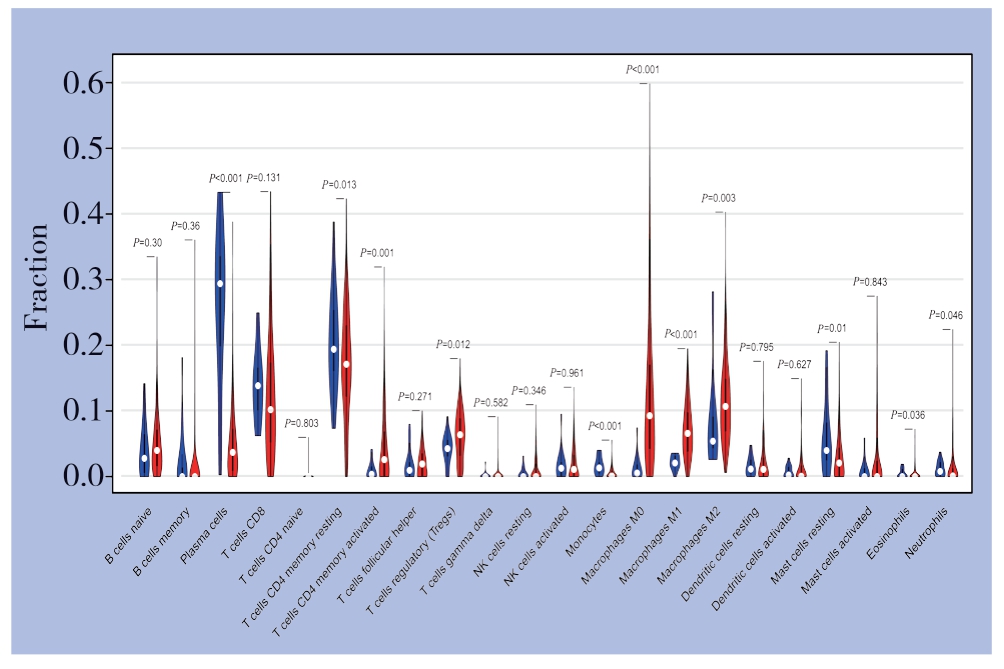
图3 GC 及正常组织中免疫细胞占比小提琴图
Figure 3 Violin diagram of the proportion of immune cells in GC and normal tissues
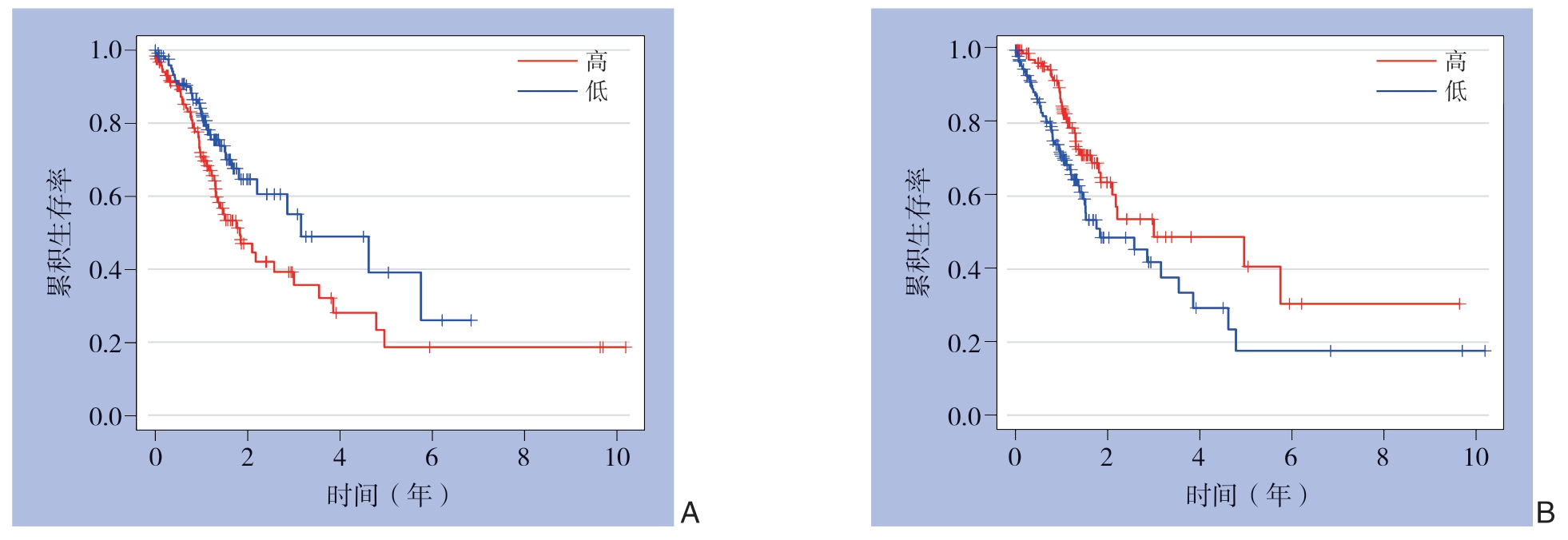
图4 免疫细胞构成水平的Kaplan-Meier 生存曲线 A:静止记忆性CD4 T 细;B:调节型T 细胞
Figure 4 Kaplan-Meier curves of different levels of immune cell composition A: T cells CD4 memory resting; B: T cells regulatory
2.3 共表达网络分析及可视化
在R语言中利用WGCNA包进行共表达网络分析及可视化。首先对从样本进行去除离群值的层次聚类绘制,并匹配相应的免疫细胞信息,从而将免疫浸润和共表达网络联系起来。结果显示,已鉴定出的两类预后相关免疫细胞,即静止记忆性CD4 T细胞和调节型T细胞与基因模块的匹配度均较好(图5)。以0.9为cutoff值,选取β软阈值为5 构建共表达网络,既保证网络接近于无尺度网络,又使其包含足够的生物信息(图6)。利用不同基因的相关性构建分层聚类树,通过动态剪接法和聚类分析,将相似的基因归类到同一模块中,最终得到5个基因模块,其中绿松石基因模块的范围最广泛(图7)。绘制热图对这5个基因模块和不同类型的免疫细胞进行相关性分析,每一行对应一个模块,每一列对应一种免疫细胞,其中每一个单元对应相关值。可以发现,绿松石模块中的基因与已鉴定出的两类预后相关免疫细胞即静止记忆性CD4 T细胞(r=0.27,P<0.001)和调节型T细胞(r=0.19,P=0.003)相关性最为显著(图8),因此将从绿松石模块中筛选这两类免疫细胞的浸润调控基因。
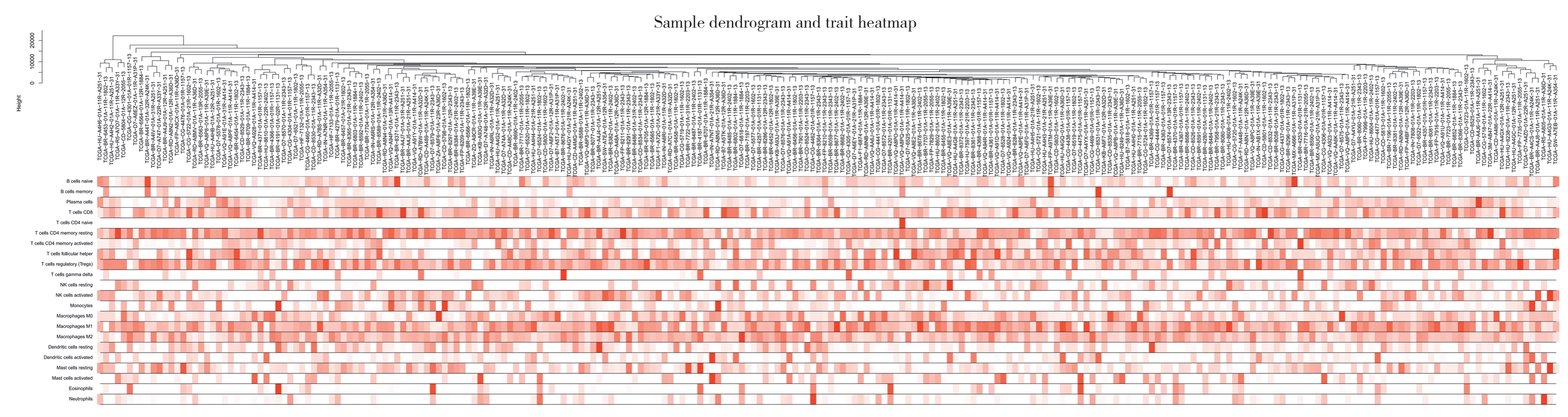
图5 样本层次聚类树结合免疫细胞表达量分析图
Figure 5 Analysis of sample level clustering combined with expression level of the immune cells
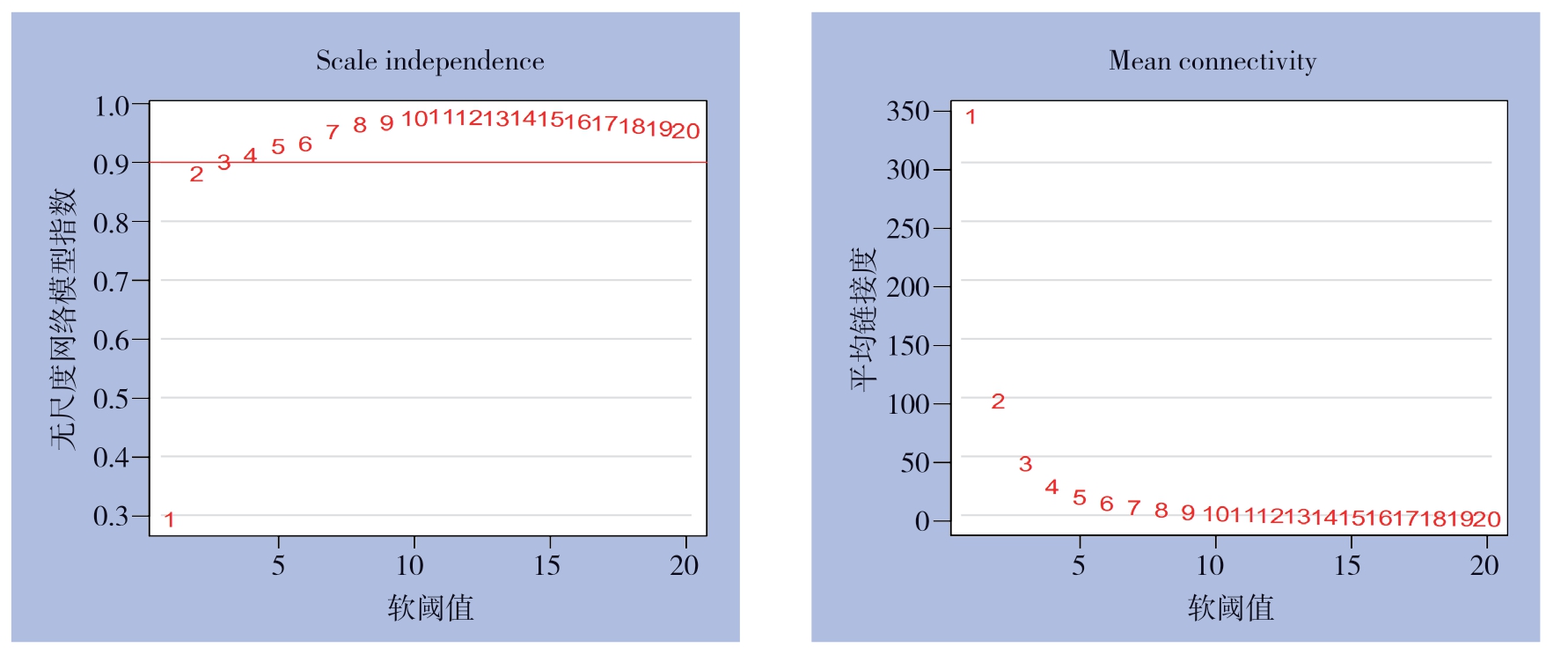
图6 WGCNA 分析软阈值(β)的确定
Figure 6 Determination of soft threshold (β) for WGCNA analysis
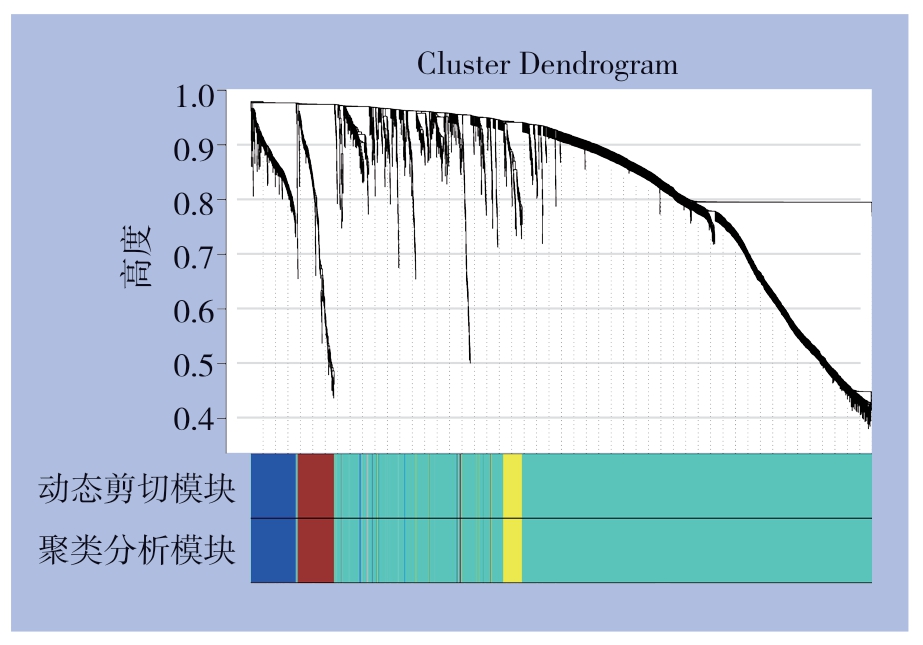
图7 GC 的共表达基因模块聚类树图
Figure 7 A cluster tree diagram of co-expressed gene modules for GC
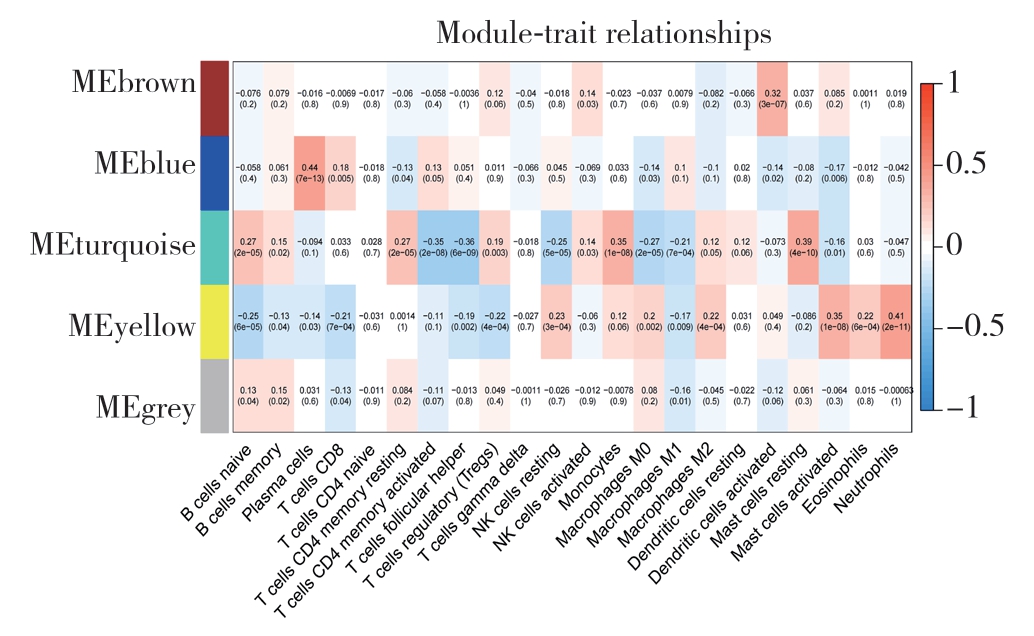
图8 热图分析模块基因与免疫细胞相关性
Figure 8 Heat map analysis of the correlation between module genes and immune cells
2.4 GC 免疫细胞浸润相关预后基因的鉴定
对与记忆性CD4 T细胞和调节型T细胞相关的绿松石模块基因进行生存分析。为了缩小与疾病无关的死亡因素的干扰,本数据集将长于10年的生存期默认为10年,并将患者的生存状态设为存活。最后共分析了355例GC患者的总生存期,共鉴定得到了3个与记忆性CD4 T细胞相关(CGB5、LINC00106、LINC00392)和一个与调节型T细胞相关(UPK1B)的生存基因(均P<0.05),其中CGB5、LINC00392、UPK1B的高表达与不良预后相关,而LINC00106的高表达则有益于患者预后(图9)。
2.5 差异生存基因的预后验证
基于在线Kaplan-Meier plotter数据库,发现CGB5、UPK1B的高表达均与GC的不良预后明显相关(P<0.05)。通过NCBI(https://www.ncbi.nlm.nih.gov)下载GSE84437,GSE57303,GSE62254等多组GEO数据库中独立数据集,验证筛选出的关键基因的预后意义,生存分析发现CGB5和UPK1B的表达与G C 患者的预后均有统计学意义(均P<0.05),证明了其具有可靠的预后相关性。然而,由于包含LINC00106和LINC00392的数据集在现行数据库中收录过少且样本数目明显不足,我们仅发现LINC00106的高表达与GC的不良预后呈负相关,但尚无统计学意义(P=0.251)。利用lnCAR(https://lncar.renlab.org),基于多组GEO数据集,发现相比于正常组织,LINC00106在GC等消化道肿瘤中均呈低表达,而LINC00392在GC等消化道肿瘤中均呈高表达,这与先前的发现一致,并在一定程度上反映了两者在GC的发生发展中发挥作用(图10)。
2.6 基因的功能富集分析
对得到的4 个预后差异基因执行包括G O 和KEGG途径在内的功能分析。GO富集分析通过对差异基因进行GO terms富集度统计学的分析,计算出差异基因FDR值(q-value),最后按照BP,MF和CC 3个层面对基因进行注释和排序。GO分析表明:这些基因主要参与的生物过程包括:肌肉系统过程、细胞器裂变、DNA−结合转录激活活性,RNA聚合酶II−特异性和糖胺聚糖结合,主要的CC有:细胞外基质和胶原蛋白细胞外基质。KEGG通路分析发现,这些基因主要富集于细胞周期和血管平滑肌收缩过程(图11)。

图9 Kaplan-Meier 法分析4 个关键基因对GC 患者预后的影响
Figure 9 Kaplan-Meier analysis of the impact of the 4 key genes on the prognosis of GC patients

图10 4 个关键基因在外部数据库的预后效能验证
Figure10 Prognostic efficacy verification of 4 key genes in external databases
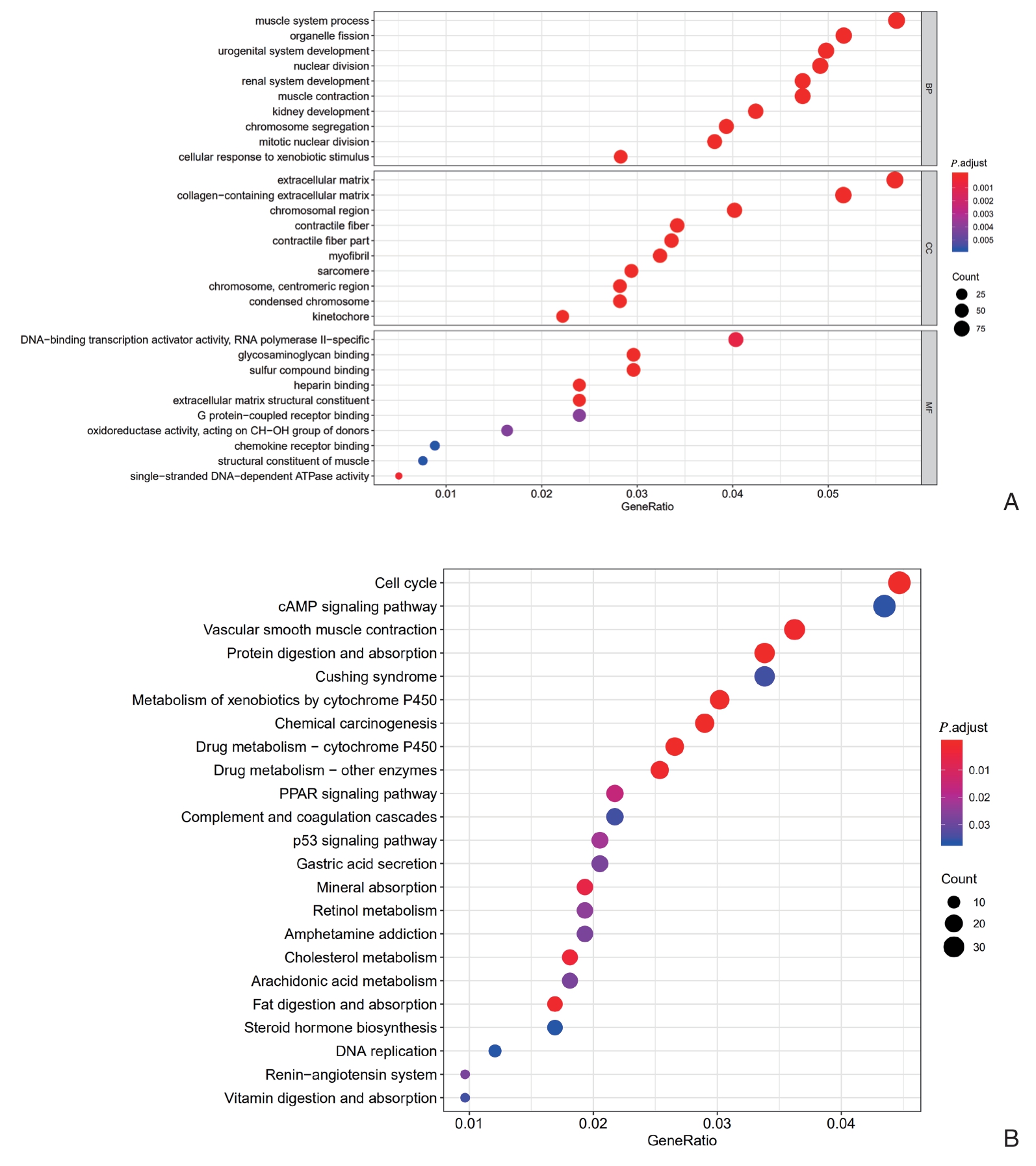
图11 预后相关基因的GO 和KEGG 功能富集分析 A:GO 途径;B:KEGG 途径
Figure 11 Functional enrichment analysis of GO and KEGG based on prognostic related genes A: GO pathway; B: KEGG pathway
3 讨 论
GC是全球范围内人类最高发的恶性肿瘤之一,但由于前期症状不明显,大部分患者确诊时肿瘤已经发展到中晚期[1],且治疗后后期复发率较高,预后效果较差,致死率居高不下[20]。近年来,肿瘤的免疫学治疗受到极大的关注[21]。GC肿瘤微环境高度复杂且异质,肿瘤相关免疫细胞在肿瘤的发生、发展、侵袭、转移中发挥作用[22],其浸润的类型和比例与患者临床结局密切相关[23]。因此探究免疫细胞相关的生物标志物对改善GC患者的预后具有重要意义[24]。
本研究首先通过CIBERSORT算法,结合TCGA数据库中患者的详细临床信息,筛选出了浸润情况与GC患者生存率呈显著相关的两类免疫细胞,即静止记忆性CD4 T细胞和调节型T细胞。静止记忆性CD4 T细胞在细胞免疫中有重要作用[25],但在肿瘤进展中的意义尚不明确,本研究发现其浸润程度与生存呈负相关,可为后续研究提供参考。对于调节型T细胞,Li等[26]研究发现其特异性转录调节因子FOXP3的高表达显示了GC良好的预后,这与本研究发现的高调节型T细胞浸润预示着GC更有利的预后一致。WGCNA作为一种高可信度的高通量数据挖掘算法,已成为鉴定疾病相关基因最可靠的方法之一。Huang等[27]基于WGCNA发现了3个与GC患者的预后相关的基因,有望将其作为GC治疗靶标。Jiang等[28]利用WGCNA构建了1个ceRNA调控网络作为膀胱癌预后标志物。然而根据对既往文献的检索发现尚未有GC方面的研究将WGCNA用于浸润免疫细胞相关预后基因的鉴定。本研究创新性地将CIBERSORT算法与WGCNA结合,基于高通量表达芯片数据,探索基因网络和免疫细胞浸润之间的关联,挖掘出了与预后免疫细胞相关性最显著的核心模块。并通过生存分析鉴定出4个GC预后相关的免疫细胞浸润调控基因(CGB5、LINC00106、LINC00392、UPK1B)。
在这些被筛选出的差异预后基因中,已有研究表明CGB5和UPK1B在肿瘤进展中发挥作用。CGB5 在滋养细胞肿瘤和某些非滋养细胞肿瘤中异常表达,可以独立预测晚期GC患者的总生存率和无复发生存率不良;并能促进卵巢癌OVCAR-3细胞血管生成拟态的形成,有望为卵巢癌的治疗提供新的靶标和思路[29-30]。UPK1B 在膀胱癌中上调,与膀胱癌的肿瘤分期,淋巴结转移,远处转移和不良预后密切相关。它通过调节Wnt/β-catenin信号传导途径,促进膀胱癌细胞的增殖、侵袭和迁移,然而目前UPK1B在GC机制中的作用尚不明确[31]。而基因LINC00106、LINC00392尚无研究显示与肿瘤相关。因此,这些基因在消化道肿瘤中的潜在作用以及它们参与的生物学功能与肿瘤之间的联系需要全面研究。
本研究回顾性地基于已经完成的国外公共数据库进行了分析,但由于患者信息获取不全,一些患有感染、免疫系统疾病或服用抗炎药的患者也有可能被纳入了这项研究,从而干扰了免疫浸润分析。此外,WGCNA算法本身在获取基因间相互作用信息时对蛋白质-蛋白质相互作用和甲基化存在依赖,也使本研究仍有一定的局限性。由于现行数据库中收录过少,本研究尚未完整地验证LINC00106和LINC00392表达与GC患者预后的关系,其作用还有待后续进一步分析鉴定。
总之,本研究基于TCGA数据库,运用合理的生物信息方法,在两类与GC生存率密切相关的免疫细胞中鉴定出了4个与GC免疫细胞浸润相关预后基因,揭示了肿瘤浸润的免疫细胞是GC预后的重要因素,为GC的早期诊断和治疗以及免疫疗法研究和新靶向药的开发提供了分子依据。
[1] Wang FH, Shen L, Li J, et al.The Chinese Society of Clinical Oncology (CSCO): clinical guidelines for the diagnosis and treatment of gastric cancer[J].Cancer Commun (Lond), 2019, 39(1):10.doi: 10.1186/s40880–019–0349–9.
[2] Bray F, Ferlay J, Soerjomataram I, et al.Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J].CA Cancer J Clin, 2018, 68(6):394–424.doi: 10.3322/caac.21492.
[3] Li Y, Wang HC, Wang JS, et al.Chemokine receptor 4 expression is correlated with the occurrence and prognosis of gastric cancer[J].FEBS Open Bio, 2020, 10(6):1149–1161.doi: 10.1002/2211–5463.12864.
[4] 刘琪, 袁波, 杜应莲.新辅助化疗联合腹腔镜手术治疗进展期胃癌的疗效及对患者预后的影响[J].中国普通外科杂志, 2019, 28(10):1302–1308.doi:10.7659/j.issn.1005–6947.2019.10.020.
Liu Q, Yuan B, Du YL.Therapeutic efficacy of neoadjuvant chemotherapy combined with laparoscopic surgical treatment of advanced stage of gastric cancer and the effect on patient prognosis[J].Chinese Journal of General Surgery, 2019, 28(10):1302–1308.doi:10.7659/j.issn.1005–6947.2019.10.020.
[5] Dashti A, Ebrahimi M, Hadjati J, et al.Dendritic cell based immunotherapy using tumor stem cells mediates potent antitumor immune responses[J].Cancer Lett, 2016, 374(1):175–185.doi: 10.1016/j.canlet.2016.01.021.
[6] Hainaut P, Plymoth A.Targeting the hallmarks of cancer: towards a rational approach to next-generation cancer therapy[J].Curr Opin Oncol, 2013, 25(1):50–51.doi: 10.1097/CCO.0b013e32835b651e.
[7] Kono K, Nakajima S, Mimura K.Current status of immune checkpoint inhibitors for gastric cancer[J].Gastric Cancer, 2020, 23(4):565–578.doi: 10.1007/s10120–020–01090–4.
[8] Xue YP, Tong LP, Liu AW, et al.Tumor-infiltrating M2 macrophages driven by specific genomic alterationsare associated with prognosis in bladder cancer[J].Oncol Rep, 2019, 42(2):581–594.doi: 10.3892/or.2019.7196.
[9] Lee DA.Cellular therapy: Adoptive immunotherapy with expanded natural killer cells[J].Immunol Rev, 2019, 290(1):85–99.doi: 10.1111/imr.12793.
[10] 邹添添, 覃伟, 朱迎, 等.肝癌免疫微环境与免疫治疗: 研究进展与发展趋势[J].中国普通外科杂志, 2020, 29(7):785–797.doi:10.7659/j.issn.1005–6947.2020.07.002.
Zou TT, Qin W, Zhu Y, et al.Immune microenvironment and immunotherapy in hepatocellular carcinoma: research progress and development directions[J].Chinese Journal of General Surgery, 2020, 29(7):785–797.doi:10.7659/j.issn.1005–6947.2020.07.002.
[11] Johnston MP, Khakoo SI.Immunotherapy for hepatocellular carcinoma: Current and future[J].World J Gastroenterol, 2019, 25(24):2977–2989.doi: 10.3748/wjg.v25.i24.2977.
[12] Newman AM, Liu CL, Green MR, et al.Robust enumeration of cell subsets from tissue expression profiles[J].Nat Methods, 2015, 12(5):453–457.doi: 10.1038/nmeth.3337.
[13] Wu YF, Zhang SJ, Yan J.IRF1 association with tumor immune microenvironment and use as a diagnosticbiomarker for colorectal cancer recurrence[J].Oncol Lett, 2020, 19(3):1759–1770.doi: 10.3892/ol.2020.11289.
[14] Xue VW, Chung JY, Córdoba CAG, et al.Transforming Growth Factor-beta: A Multifunctional Regulator of Cancer Immunity[J].Cancers (Basel), 2020, 12(11):3099.doi: 10.3390/cancers12113099.
[15] 冯道夫, 章志翔.程序性细胞死亡蛋白配体1在结直肠癌免疫治疗中的研究进展[J].中国普通外科杂志, 2020, 29(4):473–479.doi:10.7659/j.issn.1005–6947.2020.04.011.
Feng DF, Zhang ZX.Research progress of programmed death ligand 1 in immunotherapy of colorectal carcinoma[J].Chinese Journal of General Surgery, 2020, 29(4):473–479.doi:10.7659/j.issn.1005–6947.2020.04.011.
[16] Xia L, Su X, Shen J, et al.ANLN functions as a key candidate gene in cervical cancer as determined by integrated bioinformatic analysis[J].Cancer Manag Res, 2018, 10:663–670.doi: 10.2147/CMAR.S162813.
[17] AO Z, Chen Y, Lu J, et al.Identification of potential functional genes in papillary thyroid cancer by co-expression network analysis[J].Oncol Lett, 2018, 16(4):4871–4878.doi: 10.3892/ol.2018.9306.
[18] Carter SL, Brechbühler CM, Griffin M, et al.Gene coexpression network topology provides a framework for molecular characterization of cellular state[J].Bioinformatics, 2004, 20(14):2242–2250.doi: 10.1093/bioinformatics/bth234.
[19] Li A, Horvath S.Network neighborhood analysis with the multinode topological overlap measure[J].Bioinformatics, 2007, 23(2):222–231.doi: 10.1093/bioinformatics/btl581.
[20] Nie K, Shi L, Wen Y, et al.Identification of hub genes correlated with the pathogenesis and prognosis of gastric cancer via bioinformatics methods.[J].Minerva Med, 2020, 111(3):213–225.doi: 10.23736/S0026–4806.19.06166–4.
[21] Galon J, Bruni D.Daniela Tumor immunology and tumor evolution:intertwined histories[J].Immunity, 2020, 52(1):55–81.doi: 10.1016/j.immuni.2019.12.018.
[22] Griss J, Bauer W, Wagner C, et al.B cells sustain inflammation and predict response to immune checkpoint blockade in human melanoma[J].Nat Commun, 2019, 10(1):4186.doi: 10.1038/s41467–019–12160–2.
[23] Zeng D, Li M, Zhou R, et al.Tumor Microenvironment Characterization in Gastric Cancer Identifies Prognostic and Immunotherapeutically Relevant Gene Signatures[J].Cancer Immunol Res, 2019, 7(5):737–750.doi: 10.1158/2326–6066.CIR-18–0436.
[24] Wang W, He Y, Zhao Q, et al.Identification of potential key genes in gastric cancer using bioinformatics analysis[J].Biomed Rep, 2020, 12(4):178–192.doi: 10.3892/br.2020.1281.
[25] Hong GQ, Cai D, Gong JP, et al.Innate immune cells and their interaction with T cells in hepatocellular carcinoma[J].Oncol Lett, 2021, 21(1):57.doi: 10.3892/ol.2020.12319.
[26] Li FX, Sun Y, Huang JC, et al.CD4/CD8+ T cells, DC subsets, Foxp3, and IDO expression are predictive indictors of gastric cancer prognosis[J].Cancer Med, 2019, 8(17):7330–7344.doi: 10.1002/cam4.2596.
[27] Huang C, Hu GC, Ning KZ, et al.Identification of key genes controlling cancer stem cell characteristics in gastric cancer[J].World J Gastrointest Surg, 2020, 12(11):442–459.doi: 10.4240/wjgs.v12.i11.442.
[28] Jiang JZ, Bi YQ, Liu XP, et al.To construct a ceRNA regulatory network as prognostic biomarkers for bladder cancer[J].J Cell Mol Med, 2020, 24(9):5375–5386.doi: 10.1111/jcmm.15193.
[29] Yang Y, Shi Y, Hou Y, et al.CGB 5 expression is independently associated with poor overall survival and recurrence-free survival in patients with advanced gastric cancer[J].Cancer Med, 2018, 7(3):716–725.doi: 10.1002/cam4.1364.
[30] 高赛楠, 张玉泉, 苏敏, 等.CGB5对卵巢癌OVCAR-3细胞血管生成拟态形成的影响[J].肿瘤防治研究, 2016, 43(9):733–737.doi:10.3971/j.issn.1000–8578.2016.09.001.
Gao SN, Zhang YQ, Su M, et al.Effect of CGB5 on Human Epithelial Ovarian Cancer Cell Line OVCAR-3 in Vasculogenic Mimicry Formation[J].Cancer Research on Prevention and Treatment, 2016, 43(9):733–737.doi:10.3971/j.issn.1000–8578.2016.09.001.
[31] Wang F, Ma X, Xu D, et al.UPK1B promotes the invasion and metastasis of bladder cancer via regulating the Wntβ-catenin pathway[J].Eur Rev Med Pharmacol Sci, 2018, 22(17):5471–5480.doi: 10.26355/eurrev_201809_15807.
