结直肠癌(colorectal cancer,CRC)是一种全球性的常见病,是已知癌症中第三大最常见类型,占全世界癌症死亡主要原因的第二位[1]。据统计,在2018年,我国CRC新发病例数为521 590,占所有癌症新发病例的12.2%。因罹患CRC而死亡的病例数为247 563,占所有癌症病死率的8.6%[2]。而最新的统计学数据表明,预计到2021年,仅美国就将有104 270例新增CRC癌症病例及52 980例癌症死亡病例[3]。
中国大多数CRC是结肠腺癌(colon-adenocarcinoma,COAD)是发生于腺上皮细胞的恶性肿瘤,是结肠癌最主要的病理类型之一。其他罕见类型包括鳞状细胞癌、腺鳞癌、梭形细胞癌和未分化癌[4-5]。CRC是一种异质性疾病,大约60%~65%的病例是偶发的,是通过获得性体细胞遗传和表观遗传变异引起的,这些变异主要归因于可改变的潜在风险因素[6]。CRC患者的预后取决于癌症的TNM分期和是否接受治愈性手术干预,但这仅限于原发早期的CRC患者。此外,CRC患者的临床表现,治疗效果和预后还受到例如表观遗传状态和导致CRC异质性的微环境等许多因素的影响[7]。
诸多研究表明,确定能够早期进行诊断,准确监测患者病情发展及其对治疗的反应的生物标志物,对CRC患者进行早期诊断或切除是治愈大多数CRC患者的关键[8-9]。对于CRC的不良预后和高复发率,尽管最近的研究中发现的生物标志物对改善CRC的诊断和治疗做出了巨大贡献[10-14]。但由于各种原因,诸如对小型发现数据集的过度拟合和缺乏足够的临床验证之类的问题,将便捷精确的筛选生物标志物的方法纳入常规临床实践仍未实现。
近年来,由于飞速发展的微阵列技术和高通量测序技术,为寻找与癌症相关诊断、治疗和预后的关键的生物标志物提供了数据平台[15]。但由于数据的多样性及数据集之间潜在的生物学异质性和跨测量平台的技术偏见,以及研究基因表达水平所使用的传统方法需要适当的归一化,这些困难的任务为高效地利用生物信息带来了艰巨的挑战[16]。为了应对消除数据预处理(例如缩放和归一化)局限性的问题,研究人员提出了一种基于基因表达水平相对排名的方法,已在包括癌症分类在内的各种应用中均可得到可靠结果[17-19]。
研究[20-21]表明,免疫逃逸、能量异常、基因突变、促瘤炎症成为了新的肿瘤标志。肿瘤的发生和发展过程中所涉及的免疫系统的各种成分已被证明是癌症的关键性因素[22-23]。
本研究探讨一种由免疫相关基因(immunerelated gene,IRG)组成的免疫相关基因对指数(immune-related gene pair index,IRGPI)为预后标志模型的构建,并且在癌症基因组图谱计划(The Cancer Genome Atlas,TCGA)数据集和基因表达综合数据库(Gene Expression Omnibus,GEO)数据集中与CRC患者的临床信息相关联后对其进行了一系列的研究,从而证明免疫相关基因对(IRGP)与CRC患者预后的关联。
1 资料与方法
1.1 数据来源
通过公开数据进行了回顾性研究。本研究选择了2个独立数据集,包括TCGA-COAD(临床数据集452例,转录组数据集449例)以及GSE39582(585例)[24],数据收集的时间为2020年4月25日—6月20日。其中TCGA-COAD数据集因其高质量的临床记录及完善的长期随访而被用作训练数据集,其临床信息、基因表达矩阵从TCGA门户(https://portal.gdc.cancer.gov)下载。为了验证TCGA数据集中的研究结果,从GEO(https://www.ncbi.nlm.nih.gov/geo)下载了验证数据集GSE39582中的基因表达矩阵和临床数据作为外部验证。
对于TCGA-COAD及GEO数据集,基于每组注释文件将表达谱从探针水平转换为相应的基因符号,而没有进一步的标准化。对于同一个基因符号对应不同的表达值,将表达值其取均数。本研究通过对数据进行检查,仅将具有完整临床生存信息的患者用于后续分析。
1.2 CRC 特异性IRGP 的构建
进行TCGA数据集预后IRGP的构建[25]。于2020年3月28日从ImmPort数据库(https://immport.niaid.nih.gov)下载了2 498个IRG用于构建IRGP。IRG包含17类,其中包括肿瘤坏死因子家族成员、转化生长因子β家族成员、趋化因子及细胞因子受体、细胞因子、白细胞介素、干扰素等。
对本研究所涉及的TCGA及GEO平台上进行了变化相对较大(绝对偏差中值(median absolute deviation,MAD)>0.5)的IRG的测量。对每个样品中的IRG的表达值之间进行成对比较,以获得每个IRGP的得分。如果在特定IRGP中第1个IRG的表达水平高于第2个IRG,则该IRGP的得分为1。否则,分数为0。去除变异相对较小的IRGP(80%以上的数据集样本中IRGP的分数为0或1,这意味着样本之间变化很微小)后,剩下的IRGP被保留下来作为初始候选IRGP用于进行后续分析。
这种基于基因成对比较的方法具有显著优势,因为该方法是完全基于肿瘤样本的基因表达谱来计算的得分,可以不需要标准化而以个性化的方式使用[22]。
1.3 IRGPI 的构建
为得到最稳定的基因对模型来构建预后标志,将TCGA-COAD数据集以及GEO数据集中的临床数据整理,得到CRC患者样本中的生存时间及生存状态,将其与初始IRGP相合并,运用Cox回归及Kaplan-Meier法选择与CRC预后显著相关的IRGP(P<0.001)。为了避免数据过度拟合的风险,选择预后显著相关的IRGP,使用R软件包“glmnet”(版本:3.0-2)应用Lasso-Cox回归(迭代1 000次),并最终选择了20个IRGP定义为最终IRGPI。
使用R 语言包“survivalROC”(版本:1.0.3)、“survival”(版本:3.1-11)构建时间依赖性受试者工作特征(receiver operating characteristic,ROC)曲线,用以确定IRGPI的最佳截止(cut-off)值,从而将CRC患者分为低风险组和高风险组。
1.4 IRGPI 的验证
为了评估IRGPI标志的价值,首先通过Kaplan-Meier法及Log-rank检验对CRC患者绘制了生存曲线。随后选择了高-低风险组的相关临床因素,通过单变量和多变量Cox比例风险分析来评估IRGPI。在单变量分析中,针对每个数据集中的临床因素、病理因素及IRGPI进行了风险评分。随后,在多变量Cox分析中将IRGPI与可用的临床和病理变量相结合,年龄、性别及T、N分期被视为连续变量,从而进一步对IRGPI进行评估。
1.5 浸润免疫细胞的分析
采用一种可预测新鲜,冷冻及固定组织(包括实体瘤)免疫细胞浸润的流行算法,CIBERSORT[26],以便用于了解不同风险人群的免疫细胞浸润。CIBERSORT是一种从复杂组织的基因表达谱中表征其细胞组成的方法,对于紧密相关的细胞类型方面优于其他方法。CIBERSORT对肿瘤样品进行去卷积算法,对于每种细胞类型都使用了支持向量回归[27]。对于每个样品,CIBERSORT能够推断出22种浸润免疫细胞的相对比例。使用R软件包“CIBERSORT R script”(版本:1.03)对每一个样本进行了浸润免疫细胞的相对比例的计算。
1.6 功能注释和分析
为了在转录和翻译水平上了解构成IRGPI的IRG的真实表达,使用了人类蛋白质图谱(Human Protein Atlas,HPA,https://www.proteinatlas.org/)。HPA是一个可公开获得的数据库,包含近20种常见癌症的基于免疫组织化学的表达数据,并且可提供一张基于组织和器官及微阵列的免疫组化的人类组织蛋白质组图谱。从而显示了不同正常组织和癌症组织中蛋白质的空间分布[28-29]。
为了能够了解IRGPI的相关生物学功能,本研究通过R 语言包“cluster Profiler”(版本:3.14.3)对构成IRGPI的IRG进行了基因本体论(gene ontology,GO)及京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)富集分析。为了明确高风险和低风险组之间显着改变的G O 途径,在两基因组间使用Bioconductor软件包“FGSEA”(版本,1.12.0)进行基因集富集分析(Gene Set Enrichment Analysis,GSEA)[30]。本研究中涉及的生物学过程是使用了MSigDB(Molecular Signatures Database)标记基因集,选择P<0.05的数据作为有统计学意义的基因集。
1.7 统计学处理
所有统计检验均使用R 语言软件(版本3.6.1,https//www.r-proje ct.org/)及SPSS软件(SPSS version 22)进行。对于所有测试,P<0.05被认为有统计学意义。
2 结 果
2.1 IRGPI 的构建及定义
整个工作流程如图1所示。本研究共纳入有完整临床数据的1 018个样本(表1)。TCGA-COAD队列作为训练数据集,GSE39582队列作为验证数据集。从ImmPort数据库获得的2 498个IRG来构建IRGP。在所有数据集中均进行了测量,并满足训练集上的相关标准(中位绝对偏差MAD>0.5)后,共挑选出有473个IRG,并且构建了个111 392个IRGP。在训练数据集中删除相对变异较小的IRGP之后,得到12 275个IRGPs。随即合并样本临床信息,选择有统计学意义(P<0.001)的28个IRGP作为初始候选IRGP。对于初始候选IRGP使用了Lasso Cox比例风险回归并且经过了1 000次的随机循环,最终选择了20个IRGP构成IRGPI。IRGPI由28个唯一的IRG组成(表2)。
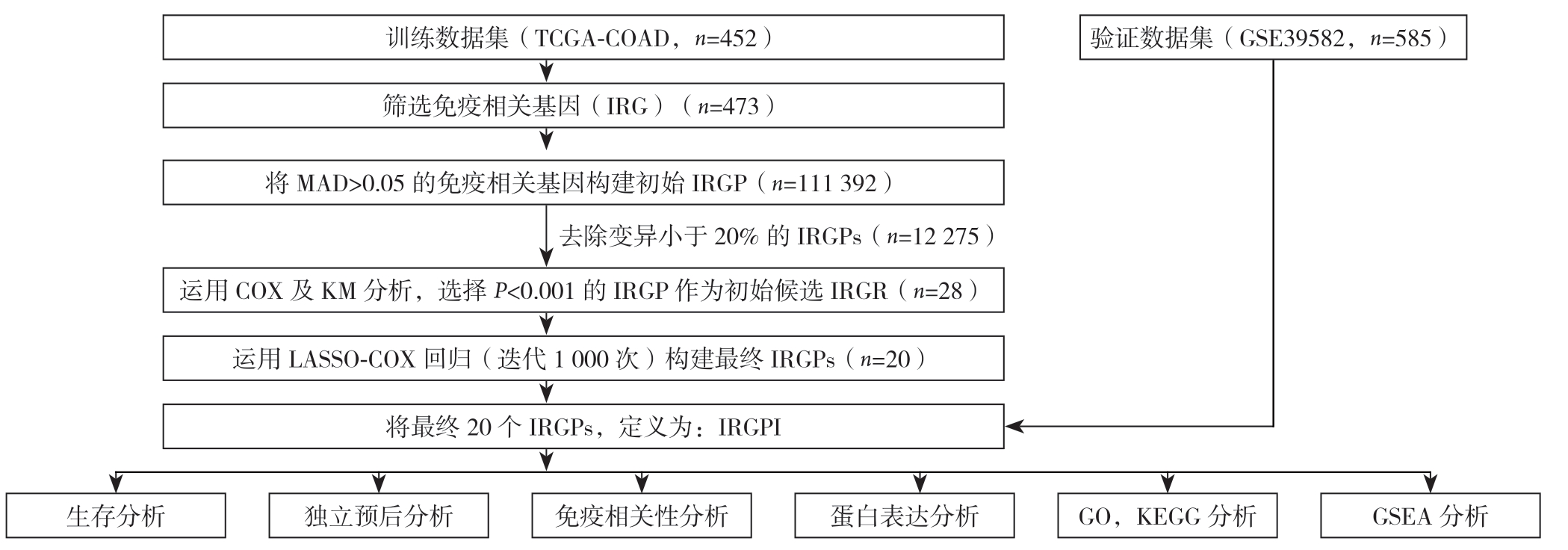
图1 构建及验证与CRC 患者IRGP 标志的工作流程
Figure 1 The workflow of constructing and verifying the IRGP signature in CRC patients
表1 TCGA-COAD 和GSE39582 数据集患者的临床特征
Table 1 Patient demographics and clinical characteristics in TCGA-COAD and GSE39582 datasets
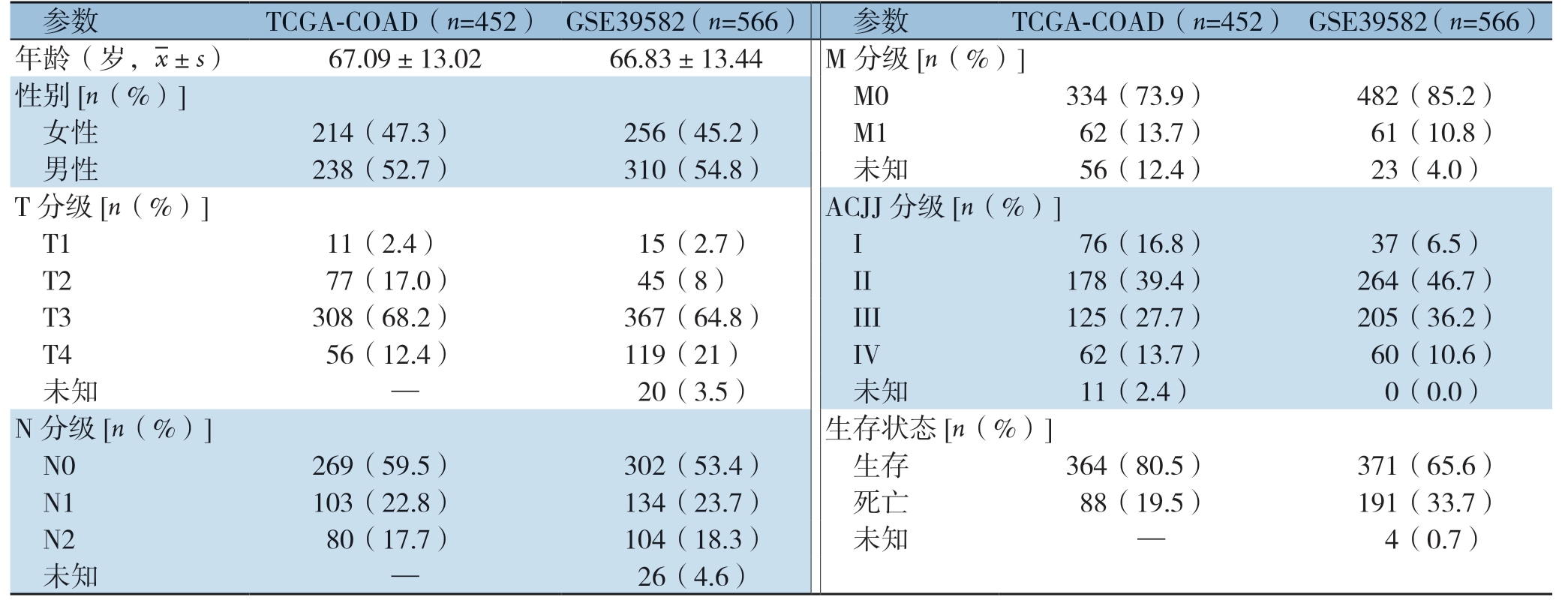
参数 TCGA-COAD(n=452) GSE39582(n=566) 参数 TCGA-COAD(n=452) GSE39582(n=566)年龄(岁,images/BZ_66_1678_802_1700_852.png±s) 67.09±13.02 66.83±13.44 M 分级[n(%)]性别[n(%)] M0 334(73.9) 482(85.2) 女性 214(47.3) 256(45.2) M1 62(13.7) 61(10.8) 男性 238(52.7) 310(54.8) 未知 56(12.4) 23(4.0)T 分级[n(%)] ACJJ 分级[n(%)] T1 11(2.4) 15(2.7) I 76(16.8) 37(6.5) T2 77(17.0) 45(8) II 178(39.4) 264(46.7) T3 308(68.2) 367(64.8) III 125(27.7) 205(36.2) T4 56(12.4) 119(21) IV 62(13.7) 60(10.6) 未知 — 20(3.5) 未知 11(2.4) 0(0.0)N 分级[n(%)] 生存状态[n(%)] N0 269(59.5) 302(53.4) 生存 364(80.5) 371(65.6) N1 103(22.8) 134(23.7) 死亡 88(19.5) 191(33.7) N2 80(17.7) 104(18.3) 未知 — 4(0.7) 未知 — 26(4.6)
表2 IRGPI 的相关信息
Table 2 Information on the IRGPI
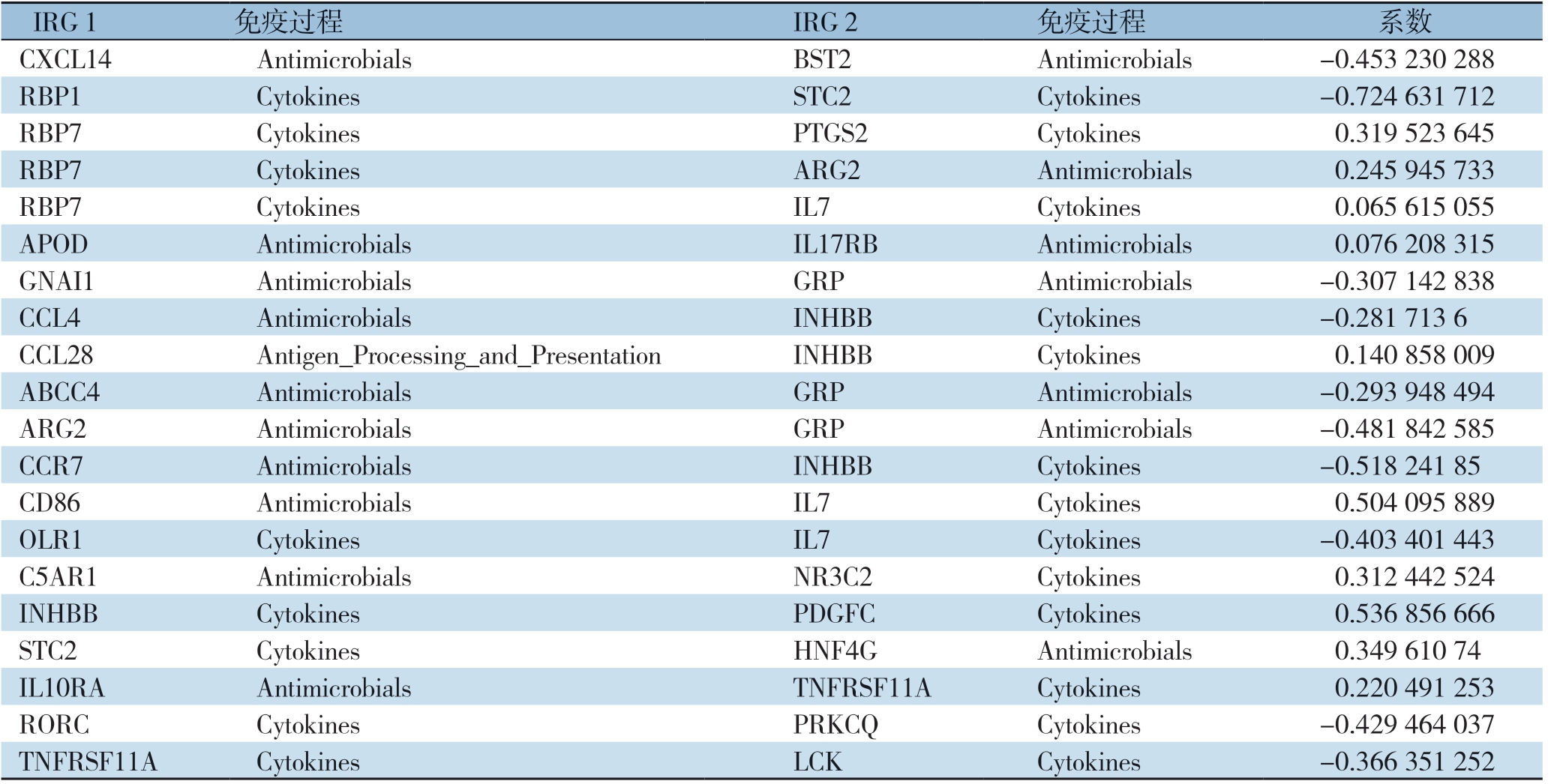
IRG 1 免疫过程 IRG 2 免疫过程 系数CXCL14 Antimicrobials BST2 Antimicrobials -0.453 230 288 RBP1 Cytokines STC2 Cytokines -0.724 631 712 RBP7 Cytokines PTGS2 Cytokines 0.319 523 645 RBP7 Cytokines ARG2 Antimicrobials 0.245 945 733 RBP7 Cytokines IL7 Cytokines 0.065 615 055 APOD Antimicrobials IL17RB Antimicrobials 0.076 208 315 GNAI1 Antimicrobials GRP Antimicrobials -0.307 142 838 CCL4 Antimicrobials INHBB Cytokines -0.281 713 6 CCL28 Antigen_Processing_and_Presentation INHBB Cytokines 0.140 858 009 ABCC4 Antimicrobials GRP Antimicrobials -0.293 948 494 ARG2 Antimicrobials GRP Antimicrobials -0.481 842 585 CCR7 Antimicrobials INHBB Cytokines -0.518 241 85 CD86 Antimicrobials IL7 Cytokines 0.504 095 889 OLR1 Cytokines IL7 Cytokines -0.403 401 443 C5AR1 Antimicrobials NR3C2 Cytokines 0.312 442 524 INHBB Cytokines PDGFC Cytokines 0.536 856 666 STC2 Cytokines HNF4G Antimicrobials 0.349 610 74 IL10RA Antimicrobials TNFRSF11A Cytokines 0.220 491 253 RORC Cytokines PRKCQ Cytokines -0.429 464 037 TNFRSF11A Cytokines LCK Cytokines -0.366 351 252
2.2 IRGPI 的验证和评估
时间相关的ROC曲线IRGPI最佳截止值为-1.239(图2)。根据CRC患者总生存率(overall survival,OS),使用IRGPI的最佳截止值将训练数据集中的患者分为低风险和高风险组(表1)。根据分析可知高风险组的OS明显低于低风险组(P=1.295×10-11,HR=6.51,95% CI= 3.79~11.21)(图3A)。
对训练数据集进行单变量和多变量Cox回归分析,以便于进一步了解IRGPI是否可以作为CRC患者的独立预后因素。在单变量Cox分析中,患者的年龄及肿瘤的T、N分期以及IRGPI对预后的影响具有统计学意义。在多变量分析中,IRGPI标记仍旧是一个独立的预后相关因素(图4A-B)。为了明确在不同人群中IRGPI是否具有与训练数据集类似的预后价值,将相同的公式应用于验证数据集(GSE39582)作为外部验证。按照与训练数据集同样的分组标准,将验证数据集的患者分为高风险和低风险组,然后进行相关生存分析后可得到与训练数据集类似的结果。高风险组的OS明显低于低风险组(P=0.000 1,HR=1.82,95% CI= 1.36~2.44)(图3B)。在验证数据集中,仍旧采用单变量和多变量Cox回归分析。最终分析结果表示:无论在单变量以及多变量Cox回归分析当中,IRGPI均有统计学意义。因此,IRGPI标记为影响CRC患者生存预后的独立因素(图4C-D)。
2.3 高、低风险组中的免疫细胞浸润
前期研究[23,31]显示,CRC细胞凋亡与其微环境中免疫细胞凋亡和浸润程度密切相关。CIBERSORT已经用于癌症微环境的相关研究中,其可以对实体肿瘤样品进行去卷积从而估计免疫细胞亚群[32-33]。本研究使用CIBERSORT来评估不同风险组中每位患者的22种不同免疫细胞的相对比例。图5A描绘了高-低风险组使用CIBERSORT输出的免疫细胞丰度的汇总,不同的风险组所浸润的免疫细胞表达量有所不同。本研究发现调节性T细胞(Tregs)(P=0.007)巨噬细胞(M0)(P=0.024)在高风险组中显着高表达,而在低风险组中静息树突状细胞(dendritic cells resting,P=0.006),静息CD4+记忆T细胞(P=1.784e-05)的百分比明显升高(图5B)。
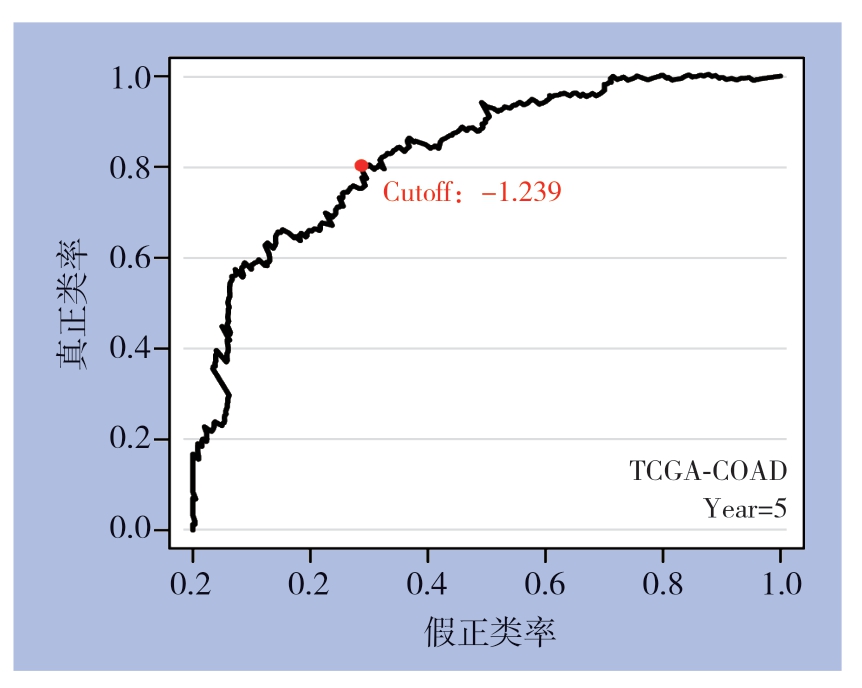
图2 训练数据集中IRGPI 的ROC 曲线
Figure 2 ROC curve of IRGPI in training dataset

图3 高、低风险组之间的CRC 患者的OS 曲线 A:训练数据集;B:验证数据集
Figure 3 OS curves of CRC patients in high-risk group and low-risk group A: Training dataset; B: Validating dataset
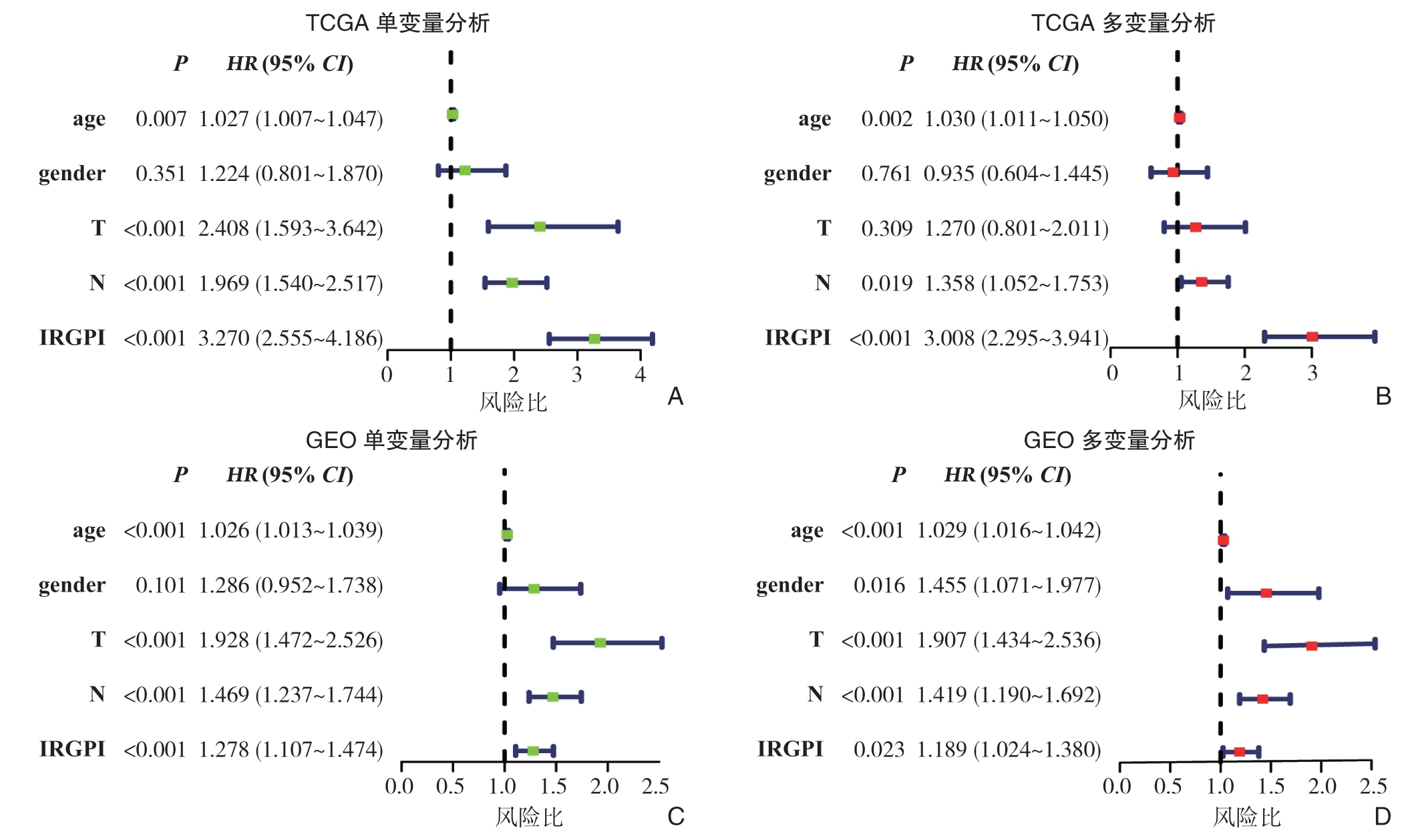
图4 CRC 患者预后的单因素和多因素Cox 回归分析的森林图 A:基于单因素分析评价IRGPI 对TCGA-COAD 中CRC 患者预后的价值;B:基于多因素分析评价IRGPI 对TCGA-COAD 中CRC 患者预后的价值;C:基于单因素分析IRGPI 对GSE39582 中CRC 患者预后的价值;D:基于多因素分析IRGPI 对GSE39582 中CRC 患者预后的价值
Figure 4 Forest plot of univariate and multivariate Cox regression analysis for the prognosis of CRC patients A: Prognostic value of IRGPI in CRC patients in TCGA-COAD based on univariate analysis; B: Prognostic value of IRGPI in CRC patients in TCGA-COAD based on multivariate analysis; C: Prognostic value of IRGPI in CRC patients in GSE39582 based on univariate analysis; D: Prognostic value of IRGPI in CRC patients in TGSE39582 based on multivariate analysis
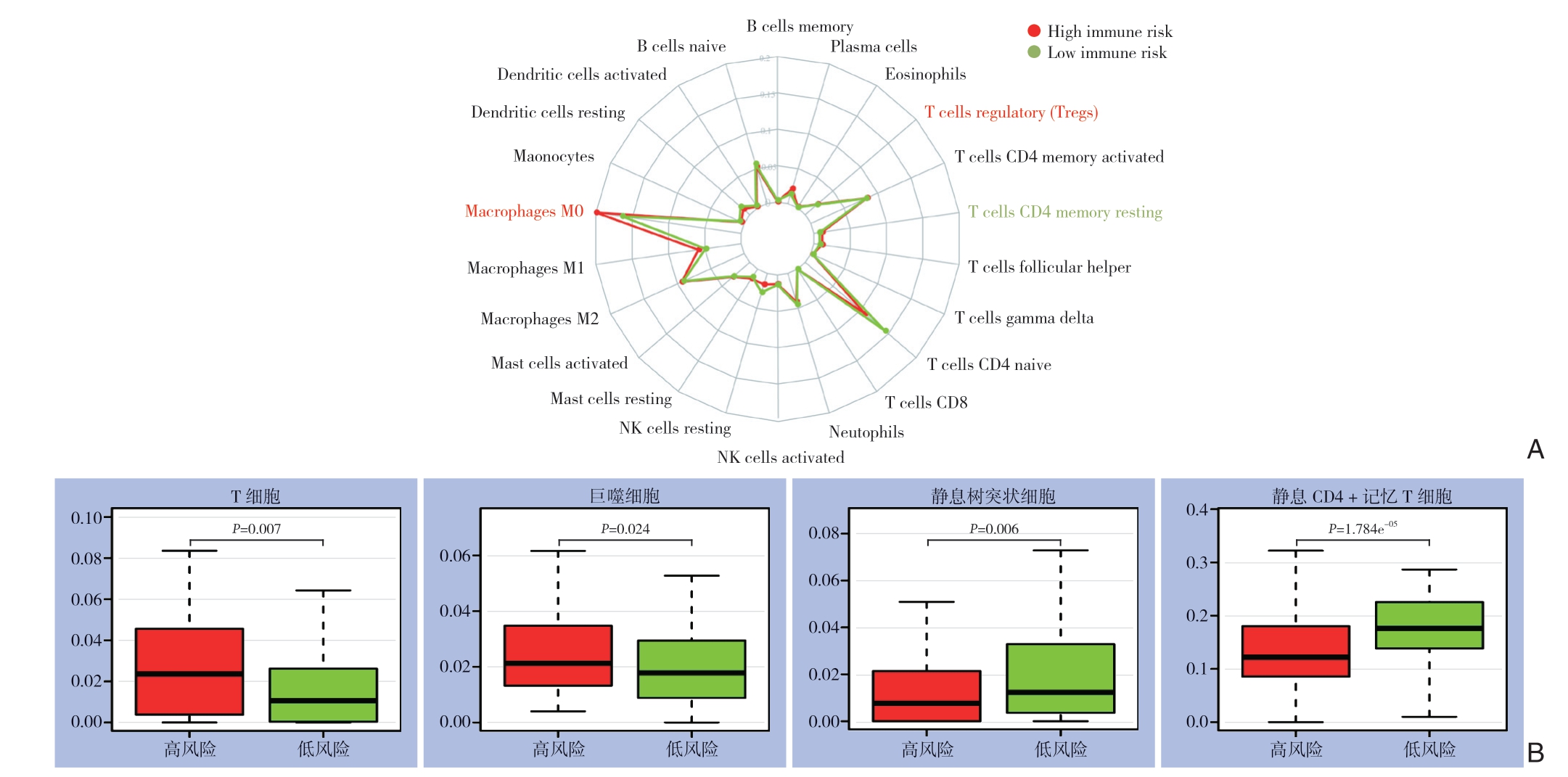
图5 CRC 患者高、低风险组中的免疫细胞浸润分析 A:CIBERSORT 评估了高、低风险组中22 种免疫细胞丰度;B:特定免疫细胞在高、低风险组中的丰度分布
Figure 5 Analysis of immunocyte infiltration in high and low risk groups of CRC patients A: CIBERSORT evaluation of the abundance of 22 immune cells in high and low risk groups; B: Abundance distribution of specific immune cells in high and low risk groups
2.4 IRGPI 的功能注释
为了验证此模型中的IRG在组织蛋白中的表达,使用HPA 数据库中免疫组织化学法分析了正常人组织和CRC组织中的蛋白质表达模式。结果表明,与正常组织比较,CRC 组织中的APOD、ARG2、BST2、CCL28、CCL4、CD86、LCK、NR3C2、PTGS2、RBP1、RBP7、STC2、TNFRSF11A的表达上调(图6)。为了解IRGPI标志所涉及的相关生物学过程,对构成IRGPI的28个IRG进行了KEGG及GO富集分析。从KEGG的分析结果显示,28个IRG所涉及的主要通路有:细胞因子-细胞因子-受体相互作用、病毒蛋白与细胞因子及细胞因子受体的相互作用、趋化因子信号通路等(图7A-B)(表2)。而GO分析表明,IRGPI中的相关IRG涉及的分子功能(MF)有:受体配体活性、细胞因子活性、趋化因子活性、细胞因子受体结合等功能。所涉及的生物过程(BP)有:白细胞迁移、细胞趋化性、白细胞-细胞黏附的调节等过程。而涉及的细胞组成(CC)大致包括:膜区、分泌颗粒膜、免疫突触等成分(图7 C)。对训练数据集的高、低风险组之间进行了GSEA分析,以研究显著改变的GO过程。基于GSEA 的分析表明,训练数据集中高-低风险组之间显著改变的生物学过程主要有:角化细胞分化、角化、表皮细胞分化、皮肤发育等过程 (图7 D-E)。与肿瘤相关的功能注释提供了IRGPI标记影响分子机制的证据,从而可用于准确预测CRC患者的预后。
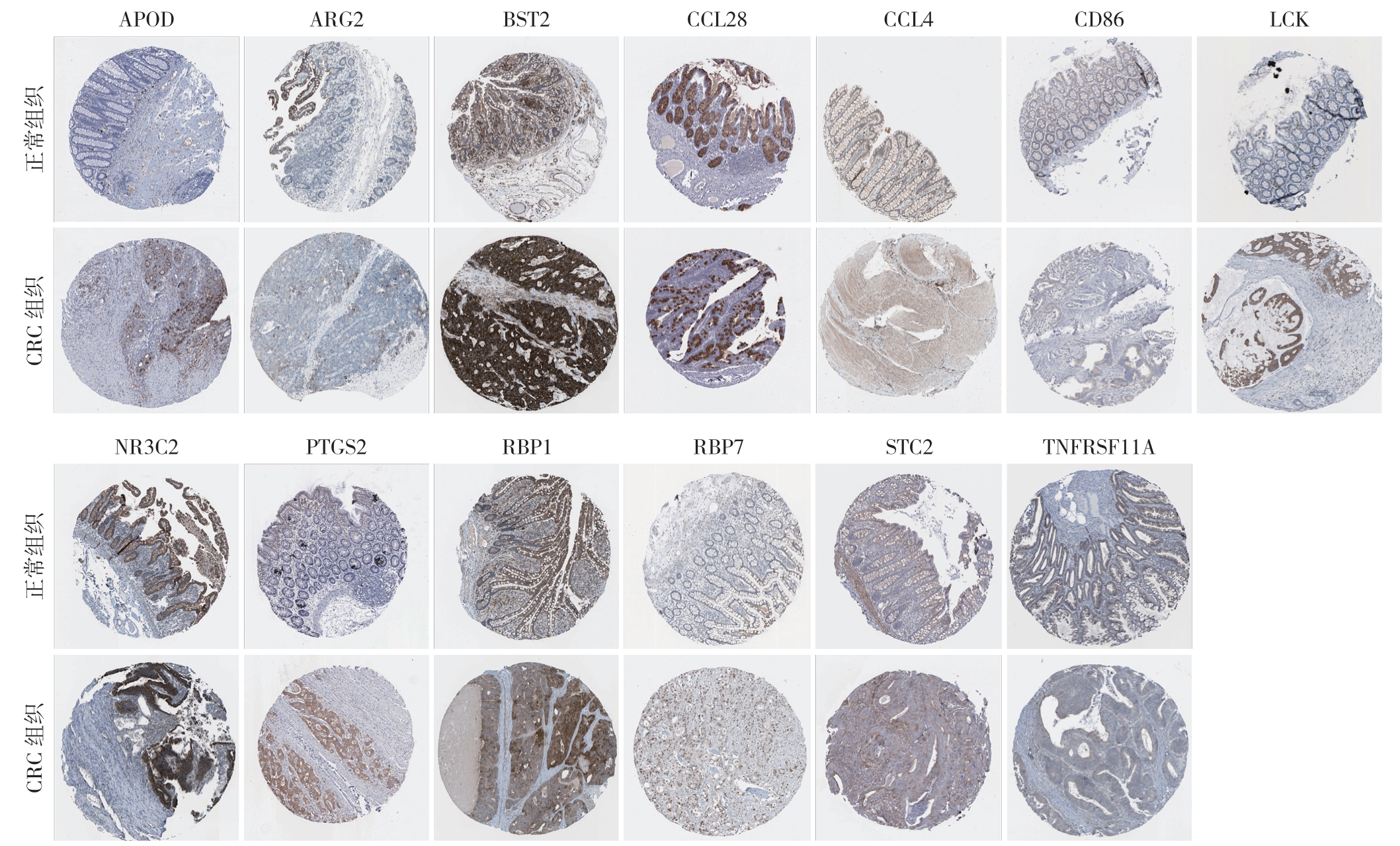
图6 CRC 组织中的蛋白质免疫组织化学法的表达(×100)
Figure 6 Expression of protein in CRC tissue by immunohistochemical method (×100)

图7 IRGPI 的功能富集分析 A-B:28 个免疫特征基因的KEGG 富集分析结果;C:28 个免疫特征基因的GO 富集分析结果; D:高、低风险组之间显著改变途径(角化,角质形成细胞分化,表皮细胞分化,皮肤发育)的GSEA 分析结果; E:28 个免疫特征基因的基因集富集分析(GSEA)结果
Figure 7 Functional enrichment Analysis of IRGPI A–B: Results of KEGG enrichment analysis of 28 immune characteristic genes; C: Results of GO enrichment analysis of 28 immune characteristic genes; D: GSEA analysis for significantly changed pathways between high and low risk groups (keratinization, epidermal cell differentiation, and skin development); E: Enrichment analysis (GSEA) 28 immune characteristic genes
3 讨 论
随着高速发展的微阵列技术和高通量测序技术,生物信息学技术成为了可以用于筛选生物标志物的强有力工具。并且在多种类癌症中得到了相关的应用及验证[34-36]。针对于高发病率的CRC,我国目前的采取以手术治疗为主,多学科综合治疗的原则。而就包括了目前正处于高速发展阶段的免疫治疗[37]。在之前有关免疫治疗的研究中所提及的:在转移性CRC患者错配修复缺陷治疗中取得明显疗效的抗程序性细胞死亡蛋白1(PD-1)药物,使得CRC的免疫治疗得到了进一步的重视[38]。为了达到增加患者生存,延缓肿瘤进展的目的,免疫治疗可通过增强机体免疫反应,激发肿瘤特异性免疫,打破免疫耐受,重新激活免疫细胞等途径从而识别并杀伤肿瘤细胞[39]。目前,比较热门的免疫治疗方式有肿瘤疫苗、免疫检查点抑制剂以及小分子治疗等,免疫治疗在一定程度上改善了CRC 患者的预后,是一种有前景的治疗选择[40]。
对于CRC的不良预后和高复发率,尽管最近的国内外研究中发现的生物标志物对改善CRC的诊断和治疗做出了巨大贡献[41-43],但是从生物的复杂性,个体差异,以及免疫因素的考虑,迫切需要找到强有力的预后生物标志物来预测CRC患者的生存情况,从而对CRC患者采取相应的治疗措施。本研究成功构建了一种由28个IRG组成的20个IRGP作为预后标志,并且在TCGA和GEO数据集中均得到了其准确性的验证,从而充分证明了IRGPI是与CRC患者预后相关的重要因素。
本研究详细分析了这些构成IRGPI的IRG,参与构建20对IRGP的大多数基因是细胞因子、抗菌剂家族成员,它们对于免疫微环境的构成中扮演者重要角色。通过HPA数据库的分析可知,CRC组织中的APOD、ARG2、BST2、CCL28、CCL4、CD86、LCK、NR3C2、PTGS2、RBP1、RBP7、STC2、TNFRSF11A基因在蛋白表达水平上较正常组织上调,表明这些基因在CRC组织中更为活跃。而在以前的研究中,提出APOD作为多种癌症类型(包括CRC)的预后标志物[44];血清中AGR2参与了卵巢交界性肿瘤病情的发生、发展[45]; BTS-2的表达增强肾细胞癌的细胞生长和侵袭性[46];胃癌组织中BST2 mRNA高表达,与肿瘤恶性程度有关,有望成为诊断胃癌新的生物标志物。BST-2 参与了人巨细胞病毒感染导致的恶性胶质瘤细胞增殖和迁移[47];骨髓来源的间充质干细胞通过CCR5促进CRC的进展[48];INHBB在CRC中过表达,并与浸润深度,淋巴结转移,远处转移和TNM分期有关,与CRC的不良OS和DFS正相关[49];肝硬化患者血清中炎性趋化因子如CCL4和CCL5的高水平表明存在肝细胞癌[50];CCR7在结肠癌组织中高表达,其表达水平与结肠癌侵袭转移密切相关[51];PTGS2表达与大肠癌患者的肿瘤复发风险增加和大肠癌特异性生存率降低有关[52];CCL28作为趋化因子,大量研究表明其在不同肿瘤中的表达异于正常组织,研究CCL28在肿瘤生成中的调控的具体作用机制有助于明确肿瘤的发生发展过程,为肿瘤的诊疗提供理论依据[53];CRBP-1过表达与舌鳞状细胞癌预后不良有关[54];RBP7的异位表达增加了结肠癌细胞的迁移和侵袭[55];STC2是CRC患者OS的独立预后因素,STC2高表达与淋巴结转移,远处转移,晚期临床阶段和较差的临床结局密切相关[56];根据前人的研究可知上述的基因参与了肿瘤发展和预后的过程,从而有助于合理的认为本研究中构建的IRGPI中的其他IRG也与CRC患者的预后相关,但仍需要进一步的实验进行相关的验证。
对于对IRG的功能注释的结果可知IRGPI中一些基因在白细胞迁移、细胞趋化性、细胞因子-细胞因子-受体相互作用等过程中起关键作用。据相关研究表明,这些标志所涉及的生物功能和途径与肿瘤的进展和预后相关[57-61]。通过GSEA指出高风险组中主要腹肌的过程有:角化细胞分化、角化、表皮细胞分化、皮肤发育等,以上途径在之前的研究中亦可证明与肿瘤的发生发展相关[62-64]。
根据对高-低风险组的免疫分析,本研究发现调节性T细胞,巨噬细胞M0在高危组中显着高表达。在过去的研究中,可知调节性T细胞,巨噬细胞M0增多,对肿瘤免疫产生负面影响,并与不良预后相关[65-68]。而对于低风险组高表达的静息树突状细胞,肿瘤浸润性树突状细胞在直肠癌组织中低表达,不成熟细胞比例增加,与TNM分期和肿瘤直径有关[22]。静息CD4+记忆T细胞与肿瘤更好的预后相关[69]。以上这些研究结果表明,IRGPI标记可作为CRC患者可靠的预后生物标记物。
本研究采用了一种新的方法用于不同平台数据的处理,相较于传统的生物标志的研究是基于全基因组水平进行的,本研究中所构建的生物标志是基于免疫相关基因而进行的,并且采取了专门设计的分析流程及方法。传统的生物信息学方法一般是根据正常或癌旁组织与肿瘤组织的基因表达差异来进行后续的研究,需要将数据进行归一化及克服跨平台的偏见,而本研究基于同一样本基因表达谱内每一个基因表达值的相对排名而进行的成对比较后得到的一组分值来构建IRGP。不仅可以避免不同数据库及平台产生的技术偏见以及因生物学异质性而带来的不同肿瘤个体间的偏差[70-71],而且也可以克服传统数据分析需要预处理数据的难点。从而得到与其他研究相比重复率较低且稳健的生物标志。本研究所使用的新方法在包括癌症在内的许多研究中均具有可靠的结果[17-19,25,72]。因此本研究所构建的生物标志可以实现单样本、精准化、高效率地评估CRC患者的预后,这是本研究所具有的优势之一。
在本研究中,IRGPI标志物的构建是使用与传统研究不同的Lasso回归经过了1 000次的迭代构建的,它可以避免过度拟合从而确定最佳的变量。利用CRC患者ROC曲线分析确定的最佳截止值,将训练数据集中的患者分为高风险组和低风险组。经过相关分析,两组预后有显著统计学差异。Lasso回归以及ROC曲线的最佳截止值的相关应用还可适用于其他不同的数据集和临床队列,这也是本研究所具有的重要优势。
本研究依旧具有局限性。首先,本研究采取的是回顾性分析,本研究所涉及的训练数据集及验证数据集均使用公开数据库中的样本。虽然两个大型公开数据库拥有公认的稳定性及可靠性,但是对于数据库中石蜡切片样品的稳定性和有效性仍有待进一步考证。如需进一步验证研究结果,则需要进行前瞻性队列研究。其次,本研究中所涉及的基因表达特征会受到由肿瘤内生物学异质性及统计学偏差所带来的影响。尽管使用了样本量较大的TCGA及GEO两个数据集,但仍旧需要包含具有不同样本属性的更多数据集,以进行更广泛的验证。最后,本研究所使用的基因表达矩阵是基于RNA-seq或微阵列平台产生的,由于其价格高,转化周期长以及对生物信息学专业知识有较高要求而使其在日常临床应用中受到了一定的限制[73]。
综上所述,本研究成功构建了由20个IRGPI标志。并且了解它们作为一种生物标志所拥有的预后价值。本研究中所构建的IRGPI标志是CRC患者的生存的独立预后因素。通过进一步生物学功能分析,可以了解这些IRGPI在CRC的发生发展中的功能,从而为CRC的治疗方式提供了一种新的思路。该标记或将成为一种评价CRC患者是否能够获益于相关免疫治疗的新型精准化预测工具。
[1] Keum N, Giovannucci E.Global burden of colorectal cancer: emerging trends, risk factors and prevention strategies[J].Nat Rev Gastroenterol Hepatol, 2019, 16(12):713–732.doi: 10.1038/s41575–019–0189–8.
[2] Feng RM, Zong YN, Cao SM, et al.Current cancer situation in China: good or bad news from the 2018 Global Cancer Statistics? [J].Cancer Commun (Lond), 2019, 39(1):22.doi: 10.1186/s40880–019–0368–6.
[3] Siegel RL, Miller KD, Fuchs HE, et al.Cancer Statistics, 2021[J].CA Cancer J Clin, 2021, 71(1):7–33.doi: 10.3322/caac.21654.
[4] Fleming M, Ravula S, Tatishchev SF, et al.Colorectal carcinoma: Pathologic aspects[J].J Gastrointest Oncol, 2012, 3(3):153–173.doi: 10.3978/j.issn.2078–6891.2012.030.
[5] 陈恩更, 陈力, 曹高扬, 等.基于免疫基因组学的结肠腺癌患者预后风险模型的建立[J].解放军医学杂志, 2020, 45(9):935–939.doi:10.11855/j.issn.0577–7402.2020.09.06.
Chen EG, Chen L, Cao GY, et al.Construction of prognostic risk model for patients with colon adenocarcinoma based on immunogenomics analysis[J].Medical Journal of Chinese People's Liberation Army, 2020, 45(9):935–939.doi:10.11855/j.issn.0577–7402.2020.09.06.
[6] Jasperson KW, Tuohy TM, Neklason DW, et al.Hereditary and familial colon cancer[J].Gastroenterology, 2010, 138(6):2044–2058.doi: 10.1053/j.gastro.2010.01.054.
[7] Linnekamp JF, Wang X, Medema JP, et al.Colorectal cancer heterogeneity and targeted therapy: a case for molecular disease subtypes[J].Cancer Res, 2015, 75(2):245–249.doi: 10.1158/0008–5472.CAN-14–2240.
[8] Kennedy RD, Bylesjo M, Kerr P, et al.Development and independent validation of a prognostic assay for stage II colon cancer using formalin-fixed paraffin-embedded tissue[J].J Clin Oncol, 2011, 29(35):4620–4626.doi: 10.1200/JCO.2011.35.4498.
[9] Ribic CM, Sargent DJ, Moore MJ, et al.Tumor microsatelliteinstability status as a predictor of benefit from fluorouracil-based adjuvant chemotherapy for colon cancer[J].N Engl J Med, 2003, 349(3):247–257.doi: 10.1056/NEJMoa022289.
[10] Cabrero-de Las Heras S, Martínez-Balibrea E.CXC family of chemokines as prognostic or predictive biomarkers and possible drug targets in colorectal cancer[J].World J Gastroenterol, 2018, 24(42):4738–4749.doi: 10.3748/wjg.v24.i42.4738.
[11] Grizzi F, Basso G, Borroni EM, et al.Evolving notions on immune response in colorectal cancer and their implications for biomarker development[J].Inflamm Res, 2018, 67(5):375–389.doi: 10.1007/s00011–017–1128–1.
[12] Ozawa T, Kandimalla R, Gao F, et al.A MicroRNA Signature Associated With Metastasis of T1 Colorectal Cancers to Lymph Nodes[J].Gastroenterology, 2018, 154(4):844–848.doi: 10.1053/j.gastro.2017.11.275.
[13] Danielsen HE, Hveem TS, Domingo E, et al.Prognostic markers for colorectal cancer: estimating ploidy and stroma[J].Ann Oncol, 2018, 29(3):616–623.doi: 10.1093/annonc/mdx794.
[14] Matsutani S, Shibutani M, Maeda K, et al.Tumor-infiltrating Immune Cells in H&E-stained Sections of Colorectal Cancer Tissue as a Reasonable Immunological Biomarker[J].Anticancer Res, 2018, 38(12):6721–6727.doi: 10.21873/anticanres.13041.
[15] Kulasingam V, Diamandis EP.Strategies for discovering novel cancer biomarkers through utilization of emerging technologies[J].Nat Clin Pract Oncol, 2008, 5(10):588–599.doi: 10.1038/ncponc1187.
[16] Leek JT, Scharpf RB, Bravo HC, et al.Tackling the widespread and critical impact of batch effects in high-throughput data[J].Nat Rev Genet, 2010, 11(10):733–739.doi: 10.1038/nrg2825.
[17] Zak DE, Penn-Nicholson A, Scriba TJ, et al.A blood RNA signature for tuberculosis disease risk: a prospective cohort study[J].Lancet, 2016, 387(10035):2312–2322.doi: 10.1016/S0140–6736(15)01316–1.
[18] Heinäniemi M, Nykter M, Kramer R, et al.Gene-pair expression signatures reveal lineage control[J].Nat Methods, 2013, 10(6):577–583.doi: 10.1038/nmeth.2445.
[19] Popovici V, Budinska E, Tejpar S, et al.Identification of a poorprognosis BRAF-mutant-like population of patients with colon cancer[J].J Clin Oncol, 2012, 30(12):1288–1295.doi: 10.1200/JCO.2011.39.5814.
[20] Mellman I, Coukos G, Dranoff G.Cancer immunotherapy comes of age[J].Nature, 2011, 480(7378):480–489.doi: 10.1038/nature10673.
[21] Hanahan D, Weinberg RA.Hallmarks of cancer: the next generation[J].Cell, 2011, 144(5):646–674.doi: 10.1016/j.cell.2011.02.013.
[22] 胡永挑, 曾小康.直肠癌组织中肿瘤浸润性树突状细胞数量和表型的改变及临床意义[J].中国普通外科杂志, 2019, 28(6):768–772.doi:10.7659/j.issn.1005–6947.2019.06.019.
Hu YT, Zeng XK.Change in number and phenotype of tumor invasive dendritic cells of rectal cancer tissue and the clinical significance[J].Chinese Journal of General Surgery, 2019, 28(6):768–772.doi:10.7659/j.issn.1005–6947.2019.06.019.
[23] 余咸静.结直肠癌细胞凋亡与其微环境中免疫细胞凋亡和浸润程度的关系[J].湖北科技学院学报: 医学版, 2021, 35(1):21–24.doi:10.16751/j.cnki.2095–4646.2021.01.0021.
Yu XJ.Relationship between Apoptosis of Colorectal Cancer Cells and Apoptosis and Infiltration Degree of Immune Cells in the Microenvironment[J].Journal of Hubei University of Science and Technology: Medical Sciences, 2021, 35(1):21–24.doi:10.16751/j.cnki.2095–4646.2021.01.0021.
[24] Marisa L, de Reyniès A, Duval A, et al.Gene expression classification of colon cancer into molecular subtypes: characterization, validation, and prognostic value[J].PLoS Med, 2013, 10(5):e1001453.doi: 10.1371/journal.pmed.1001453.
[25] Li B, Cui Y, Diehn M, et al.Development and Validation of an Individualized Immune Prognostic Signature in Early-Stage Nonsquamous Non-Small Cell Lung Cancer[J].JAMA Oncol, 2017, 3(11):1529–1537.doi: 10.1001/jamaoncol.2017.1609.
[26] Newman AM, Liu CL, Green MR, et al.Robust enumeration of cell subsets from tissue expression profiles[J].Nat Methods, 2015, 12(5):453–457.doi: 10.1038/nmeth.3337.
[27] Phillips K.Cough mixture proposal[J].JFMA, 1973, 60(5):45.
[28] Uhlen M, Zhang C, Lee S, et al.A pathology atlas of the human cancer transcriptome[J].Science, 2017, 357(6352):eaan2507.doi: 10.1126/science.aan2507.
[29] Uhlén M, Fagerberg L, Hallström BM, et al.Proteomics.Tissue-based map of the human proteome[J].Science, 2015, 347(6220):1260419.doi: 10.1126/science.1260419.
[30] Subramanian A, Tamayo P, Mootha VK, et al.Gene set enrichment analysis: a knowledge-based approach for interpreting genomewide expression profiles[J].Proc Natl Acad Sci U S A, 2005, 102(43):15545–15550.doi: 10.1073/pnas.0506580102.
[31] Liu X, Wu S, Yang Y, et al.The prognostic landscape of tumorinfiltrating immune cell and immunomodulators in lung cancer[J].Biomed Pharmacother, 2017, 95:55–61.doi: 10.1016/j.biopha.2017.08.003.
[32] Desmedt C, Salgado R, Fornili M, et al.Immune Infiltration in Invasive Lobular Breast Cancer[J].J Natl Cancer Inst, 2018, 110(7):768–776.doi: 10.1093/jnci/djx268.
[33] Ali HR, Chlon L, Pharoah PD, et al.Patterns of Immune Infiltration in Breast Cancer and Their Clinical Implications: A Gene-Expression-Based Retrospective Study[J].PLoS Med, 2016, 13(12):e1002194.doi: 10.1371/journal.pmed.1002194.
[34] 任天宇, 周新童, 党胜春.miR-486–5p的靶基因预测及其在胰腺腺癌中作用的生物信息学分析[J].中国普通外科杂志, 2020, 29(6):715–722.doi:10.7659/j.issn.1005–6947.2020.06.012.
Ren TY, Zhou XT, Dang SC.Prediction of target gene of miR-486–5p and bioinformatics analysis of their roles in pancreatic adenocarcinoma[J].Chinese Journal of General Surgery, 2020, 29(6):715–722.doi:10.7659/j.issn.1005–6947.2020.06.012.
[35] 李文菠, 孙成杰, 周国俊, 等.肝细胞癌发生发展关键基因及其功能的生物信息学分析[J].中国普通外科杂志, 2021, 30(1):32–43.doi:10.7659/j.issn.1005–6947.2021.01.005.
Li WB, Sun CJ, Zhou GJ, et al.Bioinformatics analysis of key genes and their functions in occurrence and development hepatocellular carcinoma[J].Chinese Journal of General Surgery, 2021, 30(1):32–43.doi:10.7659/j.issn.1005–6947.2021.01.005.
[36] 韩伟光, 莘玮, 苏水霞, 等.基于生物信息学的胆囊癌差异表达谱中关键蛋白调控基因分析[J].中国普通外科杂志, 2021, 30(2):165–172.doi:10.7659/j.issn.1005–6947.2021.02.006.
Han WG, Xin W, Su SX, et al.Analysis of key protein regulatory genes in differential expression profile of gallbladder cancer based on bioinformatics approaches[J].Chinese Journal of General Surgery, 2021, 30(2):165–172.doi:10.7659/j.issn.1005–6947.2021.02.006.
[37] 张宇涛.结直肠癌的治疗进展[J].山西医药杂志, 2020, 49(5):535–536.doi:10.3969/j.issn.0253–9926.2020.05.012.
Zhang YT.Progress in treatment of colorectal cancer[J].Shanxi Medical Journal, 2020, 49(5):535–536.doi:10.3969/j.issn.0253–9926.2020.05.012.
[38] 冯道夫, 章志翔.程序性细胞死亡蛋白配体1在结直肠癌免疫治疗中的研究进展[J].中国普通外科杂志, 2020, 29(4):473–479.doi:10.7659/j.issn.1005–6947.2020.04.011.
Feng DF, Zhang ZX.Research progress of programmed death ligand 1 in immunotherapy of colorectal carcinoma[J].Chinese Journal of General Surgery, 2020, 29(4):473–479.doi:10.7659/j.issn.1005–6947.2020.04.011.
[39] 邹添添, 覃伟, 朱迎, 等.肝癌免疫微环境与免疫治疗:研究进展与发展趋势[J].中国普通外科杂志, 2020, 29(7):785–797.doi:10.7659/j.issn.1005–6947.2020.07.002.
Zou TT, Qin W, Zhu Y, et al.Immune microenvironment and immunotherapy in hepatocellular carcinoma: research progress and development directions[J].Chinese Journal of General Surgery, 2020, 29(7):785–797.doi:10.7659/j.issn.1005–6947.2020.07.002.
[40] 曾家兴, 丁杰, 夏宇, 等.结直肠癌免疫治疗的研究进展[J].中国癌症防治杂志, 2020, 12(6):691–695.doi:10.3969/j.issn.1674–5671.2020.06.17.
Zeng JX, Ding J, Xia Y, et al.Research Progress of Immunotherapy for Colorectal Cancer[J].Chinese Journal of Oncology Prevention and Treatment, 2020, 12(6):691–695.doi:10.3969/j.issn.1674–5671.2020.06.17.
[41] Sveen A, Kopetz S, Lothe RA.Biomarker-guided therapy for colorectal cancer: strength in complexity[J].Nat Rev Clin Oncol, 2020, 17(1):11–32.doi: 10.1038/s41571–019–0241–1.
[42] 丁志祥, 史兵伟, 王永仿, 等.基于生物信息学对结肠癌潜在枢纽基因的筛选及验证[J].中国免疫学杂志, 2021, 37(2):201–205.doi:10.3969/j.issn.1000–484X.2021.02.014.
Ding ZX, Shi BW, Wang YF, et al.Screening and validation of potential hub genes in colon cancer based on bioin-formatics analysis[J].Chinese Journal of Immunology, 2021, 37(2):201–205.doi:10.3969/j.issn.1000–484X.2021.02.014.
[43] 胡清荣, 李瑾.基于生物信息学分析结直肠癌诊断标志物与治疗靶点[J].中国肛肠病杂志, 2020, 40(10):1–3.
Hu QR, Li J.An Analysis on Diagnostic Marks and Treatment Targets of Colorectal Cancer in View of Patients'Biological Informatics[J].Chinese Journal of Coloproctology, 2020, 40(10):1–3.
[44] Bajo-Grañeras R, Crespo-Sanjuan J, García-Centeno RM, et al.Expression and potential role of apolipoprotein D on the deathsurvival balance of human colorectal cancer cells under oxidative stress conditions[J].Int J Colorectal Dis, 2013, 28(6):751–766.doi: 10.1007/s00384–012–1616–2.
[45] 罗磊, 龚宝成, 刘福囝.GSE74602芯片数据中直肠癌关键基因与治疗药物的生物信息学筛选[J].中国普通外科杂志, 2018, 27(4):457–467.doi:10.3978/j.issn.1005–6947.2018.04.011.
Luo L, Gong BC, Liu FN.Bioinformatics screening for pivotal genes and therapeutic drugs of colorectal cancer in GSE74602 microarray data[J].Chinese Journal of General Surgery, 2018, 27(4):457–467.doi:10.3978/j.issn.1005–6947.2018.04.011.
[46] Pham QT, Oue N, Yamamoto Y, et al.The Expression of BTS-2 Enhances Cell Growth and Invasiveness in Renal Cell Carcinoma[J].Anticancer Res, 2017, 37(6):2853–2860.doi: 10.21873/anticanres.11637.
[47] 王淋淋, 李莹莹, 王语, 等.BST-2参与HCMV感染诱导的恶性胶质瘤细胞增殖和迁移[J].微生物学杂志, 2018, 38(2):77–84.doi:10.3969/j.issn.1005–7021.2018.02.012.
Wang LL, Li YY, Wang Y, et al.Protein BST-2 Involved in HCMV Infection-Induced Proliferation and Migration in Malignant glioma cells[J].Journal of Microbiology, 2018, 38(2):77–84.doi:10.3969/j.issn.1005–7021.2018.02.012.
[48] Nishikawa G, Kawada K, Nakagawa J, et al.Bone marrow-derived mesenchymal stem cells promote colorectal cancer progression via CCR5[J].Cell Death Dis, 2019, 10(4):264.doi: 10.1038/s41419–019–1508–2.
[49] Yuan J, Xie A, Cao Q, et al.INHBB Is a Novel Prognostic Biomarker Associated with Cancer-Promoting Pathways in Colorectal Cancer[J].Biomed Res Int, 2020, 2020:6909672.doi: 10.1155/2020/6909672.
[50] Sadeghi M, Lahdou I, Oweira H, et al.Serum levels of chemokines CCL4 and CCL5 in cirrhotic patients indicate the presence of hepatocellular carcinoma[J].Br J Cancer, 2015, 113(5):756–762.doi: 10.1038/bjc.2015.227.
[51] 李高勤, 雍文兴, 牛帆, 等.结肠癌免疫相关基因对预后模型构建及预后的相关性分析[J].生物医学工程与临床, 2021, 25(2):210–218.doi: 10.13339/j.cnki.sglc.20210226.017.
Li GQ, Yong WX, Niu F, et al.Construction of prognosis model and correlation analysis between immune-related gene pairs and prognosis in colon cancer[J].Biomedical Engineering and Clinical Medicine, 2021, 25(2):210–218.doi: 10.13339/j.cnki.sglc.20210226.017.
[52] Kunzmann AT, Murray LJ, Cardwell CR, et al.PTGS2 (Cyclooxygenase-2) expression and survival among colorectal cancer patients: a systematic review[J].Cancer Epidemiol Biomarkers Prev, 2013, 22(9):1490–1497.doi: 10.1158/1055–9965.EPI-13–0263.
[53] 梁天, 朱晓明.趋化因子CCL28研究进展及其与肿瘤的关系[J].陕西医学杂志, 2019, 48(6):813–814.doi:10.3969/j.issn.1000–7377.2019.06.037.
Liang T, Zhu XM.Research progress of chemokine CCL28 and its relationship to cancer[J].Shaanxi Medical Journal, 2019, 48(6):813–814.doi:10.3969/j.issn.1000–7377.2019.06.037.
[54] Chen Y, Tian T, Mao MJ, et al.CRBP-1 over-expression is associated with poor prognosis in tongue squamous cell carcinoma[J].BMC Cancer, 2018, 18(1):514.doi: 10.1186/s12885–018–4249–1.
[55] Elmasry M, Brandl L, Engel J, et al.RBP7 is a clinically prognostic biomarker and linked to tumor invasion and EMT in colon cancer[J].J Cancer, 2019, 10(20):4883–4891.doi: 10.7150/jca.35180.
[56] Li Q, Zhou X, Fang Z, et al.Effect of STC2 gene silencing on colorectal cancer cells[J].Mol Med Rep, 2019, 20(2):977–984.doi: 10.3892/mmr.2019.10332.
[57] D'agostino G, Cecchinato V, Uguccioni M.Chemokine Heterocomplexes and Cancer: A Novel Chapter to Be Written in Tumor Immunity [J].Front Immunol, 2018, 9:2185.doi: 10.3389/fimmu.2018.02185.
[58] Lin P, Guo YN, Shi L, et al.Development of a prognostic index based on an immunogenomic landscape analysis of papillary thyroid cancer[J].Aging (Albany NY), 2019, 11(2):480–500.doi: 10.18632/aging.101754.
[59] Jiang SH, Zhu LL, Zhang M, et al.GABRP regulates chemokine signalling, macrophage recruitment and tumour progression in pancreatic cancer through tuning KCNN4-mediated Ca(2+) signalling in a GABA-independent manner[J].Gut, 2019, 68(11):1994–2006.doi: 10.1136/gutjnl-2018–317479.
[60] Ocana A, Nieto-Jiménez C, Pandiella A, et al.Neutrophils in cancer: prognostic role and therapeutic strategies[J].Mol Cancer, 2017, 16(1):137.doi: 10.1186/s12943–017–0707–7.
[61] Watts TL, Cui R, Szaniszlo P, et al.PDGF-AA mediates mesenchymal stromal cell chemotaxis to the head and neck squamous cell carcinoma tumor microenvironment[J].J Transl Med, 201, 14(1):337.doi: 10.1186/s12967–016–1091–6.
[62] Goñi-de-cerio F, Thevenot J, Oliveira H, et al.Cellular Uptake and Cytotoxic Effect of Epidermal Growth Factor Receptor Targeted and Plitidepsin Loaded Co-Polymeric Polymersomes on Colorectal Cancer Cell Lines[J].J Biomed Nanotechnol, 2015, 11(11):2034–2049.doi: 10.1166/jbn.2015.2148.
[63] Meyer SN, Galván JA, Zahnd S, et al.Co-expression of cytokeratin and vimentin in colorectal cancer highlights a subset of tumor buds and an atypical cancer-associated stroma[J].Hum Pathol, 2019, 87:18–27.doi: 10.1016/j.humpath.2019.02.002.
[64] Bottomley MJ, Thomson J, Harwood C, et al.The Role of the Immune System in Cutaneous Squamous Cell Carcinoma[J].Int J Mol Sci, 2019, 20(8):2009.doi: 10.3390/ijms20082009.
[65] Conway EM, Pikor LA, Kung SH, et al.Macrophages, Inflammation, and Lung Cancer[J].Am J Respir Crit Care Med, 2016, 193(2):116–130.doi: 10.1164/rccm.201508–1545CI.
[66] Shang B, Liu Y, Jiang SJ, et al.Prognostic value of tumorinfiltrating FoxP3+ regulatory T cells in cancers: a systematic review and meta-analysis[J].Sci Rep, 2015, 5:15179.doi: 10.1038/srep15179.
[67] Ngambenjawong C, Gustafson HH, Pun SH.Progress in tumorassociated macrophage (TAM)-targeted therapeutics[J].Adv Drug Deliv Rev, 2017, 114:206–221.doi: 10.1016/j.addr.2017.04.010.
[68] Takeuchi Y, Nishikawa H.Roles of regulatory T cells in cancer immunity[J].Int Immunol, 2016, 28(8):401–409.doi: 10.1093/intimm/dxw025.
[69] 李炜, 曾静媛, 罗波,等.膀胱癌患者高表达活化的CD4+记忆性T细胞和CD8+T细胞和低表达M0巨噬细胞与临床预后佳有关[J].细胞与分子免疫学杂志, 2020, 36(2):97–103.
Li W, Zeng JY, Luo B, et al.High expression of activated CD4(+) memory T cells and CD8(+) T cells and low expression of M0 macrophage are associated with better clinical prognosis in bladder cancer patients[J].Chinese Journal of Cellular and Molecular Immunology, 2020, 36(2):97–103.
[70] Eddy JA, Sung J, Geman D, et al.Relative expression analysis for molecular cancer diagnosis and prognosis[J].Technol Cancer Res Treat, 2010, 9(2):149–159.doi: 10.1177/153303461000900204.
[71] Tan AC, Naiman DQ, Xu L, et al.Simple decision rules for classifying human cancers from gene expression profiles[J].Bioinformatics, 2005, 21(20):3896–3904.doi: 10.1093/bioinformatics/bti631.
[72] Eng PL, Zhou XY, Yi GD, et al.Identification of a novel gene pairs signature in the prognosis of gastric cancer[J].Cancer Med, 2018, 7(2):344–350.doi: 10.1002/cam4.1303.
[73] Wu J, Zhao Y, Zhang J, et al.Development and validation of an immune-related gene pairs signature in colorectal cancer[J].Oncoimmunology, 2019, 8(7):1596715.doi: 10.1080/2162402X.2019.1596715.
